第一作者简介:高林林,女,山西吕梁人,硕士,副主任法医师,研究方向为法医物证学。E-mail: myhappyfamily_gao@163.com
本文验证前期已构建的基于DNA甲基化的9-CpG年龄推断模型并对其进行优化,进一步拓展年龄推断模型的应用范围。采集浙江汉族个体血液制备成血斑样本,男性100份,女性96份。男、女性样本年龄范围均为16~65岁,年龄均值分别为(41±13)岁、(38±13)岁,并按照年龄段各分为5组。磁珠法提取DNA后采用重亚硫酸盐转化,使用EpiTYPER技术平台检测模型中的9个CpG甲基化位点,最后按照多元回归推断公式计算年龄值。结果表明,所有样本的转化DNA平均回收率为42.92%±14.03%。男性样本的推断年龄与实际年龄的平均绝对偏差(MAD)为2.95岁,不同年龄组间的MAD值无明显差异,年龄高估与低估的样本数基本相等;女性样本的MAD为3.18岁,不同年龄组间的MAD差异显著。对女性样本调整回归模型后其年龄推断的MAD为2.65岁,不同年龄组间的MAD值较为均一。前期已构建的9-CpG年龄推断模型适用于浙江男性汉族个体的年龄推断,调整优化后的年龄推断模型对浙江女性汉族个体的预测更为精准,且上述两个预测模型均适用于现场血斑样本。
The DNA methylation-based 9-CpG age prediction model was to verify and optimize with purpose to further expand its application scope. From 100 male and 96 female individuals of Zhejiang Han-ethnic population, the blood samples were collected and left to separately dry on gauze. The male and female blood samples were divided into 5 groups according to their ages (ranging from 16—65 years old), having their DNA extracted with magnetic bead method and then converted through bisulfite. EpiTYPER platform was adopted to detect the nine methylation sites available with the model. The predicted age value was calculated under the multiple regression inference formula. The average recovery of converted genome DNA was 42.92%±14.03%. For male samples, the mean absolute deviation (MAD) was about 2.95 years between the predicted age and the real, having no significant difference in MAD values between the age-different groups but showing basically equal about the number of samples relating to age over-/under-estimation. For female samples, the MAD was 3.18 years, quite different between discrepant age groups and able to reduce to 2.65 years after the regression model was adjusted to result in relative similarity among different age groups. The DNA methylation-based 9-CpG age model established previously is suitable for prediction of Zhejiang Han-ethnic male individuals with detection of relevant bloodstains collected on the crime scene, and capable of being used for Zhejiang Han-ethnic female individuals with bloodstain as the samples after implementation of adjustment and optimization.
个体的年龄推断一直是法医学领域的研究热点, 通过骨骼、牙齿等推断个体年龄的方法[1, 2], 无法用于实际案件中遇到的各种组织及体液供者的年龄推断; 通过端粒长度变化来推断年龄的定量研究尚在摸索阶段[3]; 通过DNA甲基化水平来推断个体年龄的研究在国内外均已有广泛开展[4, 5, 6, 7]。本研究组前期通过不同年龄段的北方男性血液样本建立了DNA甲基化水平的年龄推断模型, 通过质谱方法可检测9个CpG位点的甲基化值, 该9-CpG年龄推断模型与同类研究相比, 不仅误差值较小, 且已有实战应用案例[8, 9]。但由于原计算模型针对的是河北、北京、河南等北方汉族男性, 对于如浙江等我国南部地区的人群, 其生活习性和遗传背景与北方汉族个体有显著的差异, 能否使用该模型推断年龄尚需验证。随着全国Y染色体STR数据库的不断建设, 在庞大复杂的家系排查中, 若能推断出现场遗留生物检材所属个体的年龄, 则可大幅提升排查工作效率。因此本研究通过检测血斑样本, 采用上述模型推断浙江汉族个体的年龄, 希冀验证并拓展9-CpG年龄推断模型的适用性及范围。
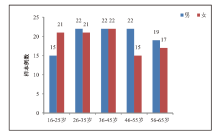
根据知情同意原则, 采集浙江汉族男性及女性无关个体新鲜外周血样本共196份(男100份, 女96份), 采样经公安部鉴定中心科研伦理委员会审核批准。每份取约600 μL血液滴于三层折叠的白纱布上制成血斑纱布, 阴干后室温保存3~6个月。实战中涉案人员的年龄分布大多集中在16~65岁, 故采样选取此年龄段个体且尽量每个年龄选取2人份, 各年龄段样本保持均等。样本年龄段分组具体见图1。
MagAttract® M48 DNA Manual 试剂盒(Qiagen公司, 德国); EZ DNA MethylationTM试剂盒(Zymo Research公司, 美国); QuantStudio5荧光定量PCR仪、Quantifiler® Trio DNA定量试剂盒、NanoDrop 2000c分光光度计均购自美国Thermo Scientific公司; Eppendorf Pro S PCR扩增仪(Eppendorf公司, 德国); 虾碱性磷酸酶(Shrimp Alkaline Phosphatase, SAP)、MassARRAY® Clean Resin、MassARRAY质谱仪及EpiTYPER v1.3软件均购自美国Agena Bioscience公司。
剪取三层折叠血纱布上血斑约2.0 cm×2.0 cm, 采用MagAttract® M48 DNA Manual试剂盒按照说明书提取基因组DNA, 洗脱体积为60 μL。采用NanoDrop 2000c分光光度计测定DNA浓度及纯度, 所有DNA模板保存至-20 ℃备用。
剪取男性及女性各13份样本血纱布上血斑约1.0 cm×1.0 cm, 采用1.3所述方法进行DNA提取。取1 μL DNA模板采用NanoDrop 2000c分光光度计测定DNA质量; 取2 μL DNA模板采用Quantifiler® Trio DNA定量试剂盒以QuantStudio5实时荧光定量PCR仪按照试剂盒说明书进行DNA质量测定。
取1 μg DNA样本, 若总量不足1 μg, 则取45 μL DNA样本, 按照EZ DNA MethylationTM试剂盒说明书进行重亚硫酸盐处理, 转化后的DNA采用30 μL纯水洗脱, 并再次采用NanoDrop 2000c分光光度计测定转化后的DNA质量。
9对扩增引物均按照文献[8]进行合成。扩增体系为5 μL, 其中10×PCR 缓冲液0.5 μL, dNTP 0.04 μL, 5U PCR 聚合酶0.1 μL, 2 μmol/L 引物2.0 μL, 转化后DNA(bisulfite converted DNA, BS-DNA)2.36 μL。与文献方法[10]相比, 引物浓度由1 μmol/L增加到2 μmol/L, PCR 聚合物从0.09 μL增加到0.1 μL。扩增程序及SAP纯化均按照文献[10]进行。
转录及T裂解体系为5 μL, 按文献[10]配比混匀, 加入SAP纯化后产物4 μL, 37 ℃孵育3 h。4 ℃存储。
96孔PCR板的每孔内加入36 μL不含RNA酶的水以及T裂解产物9 μL, 混匀后离心15 min, 质谱仪的树脂盒内加入适量纯化树脂, 并将芯片放置于相应位置, 采用MassARRAY系统进行检测。
转化回收率计算方法:转化后得到的单链DNA总量与转化前加入的双链基因组DNA总量的百分比值。
所有甲基化值采用EpiTYPER v1.3软件进行数据处理, 并按照已建立的线性回归方程[8]进行个体年龄预测计算。将预测结果与人员实际年龄进行比对, 计算个体预测年龄与实际年龄差值绝对值的平均值即平均绝对偏差(mean absolute deviation, MAD)值; 计算个体预测年龄与实际年龄差值的平均值即平均误差(mean error, ME), 正值表示高估、负值表示低估, 评估已建模型[8]对本研究样本的适用性。采用R软件 “lm”函数建立多元线性回归模型, 采用IBM SPSS Statistics 23对所有处理数据作t检验、单因素方差分析等。
26份样本同时用两种不同的定量方法进行DNA质量测定后发现, NanoDrop 2000c分光光度计所测定的DNA质量均值为(9.15±4.45)ng/μL, 而实时荧光PCR定量法获得的DNA质量均值为(3.79±2.80) ng/μL, 两者之间具有显著性差异(P< 0.05)。前者所测定的DNA量均值是后者均值的2.4倍。
所有样本提取及转化后的DNA定量结果(NanoDrop 2000c分光光度计测定)、转化回收率见表1。不同样本提取得到的DNA及转化后得到的DNA总量存在显著性差异, 样本间的甲基化转化回收率也有明显差异。血斑提取得到的DNA浓度最低为11.90 ng/μL, 最高可达205.70 ng/μL, 平均OD值(按A260/A280测算)为1.69±0.16; 进行重亚硫酸盐转化的DNA总量最低为535.50 ng(11.90 ng/μL×45 μL), 重亚硫酸盐转化后DNA(bisulfite-converted DNA, BS-DNA)总量最低为153.00 ng(5.10 ng/μL×30 μL)。
| 表1 不同样本提取与转化后DNA定量以及转化回收率的比较 Table 1 Quantity range of extracted genome DNA, bisulfite-converted DNA and recovery rates |
为进一步分析最低的基因组DNA用量, 以最终PCR扩增所需的BS-DNA量结合转化回收率进行计算。BS-DNA最低浓度为5.10 ng/μL, 每个PCR反应加入2.36 μL, 共7个PCR反应, 因此最低需要BS-DNA 84.00 ng。按照平均转化回收率43%计算, 最低的基因组DNA用量为195.00 ng, 如果按照最高转化回收率计算, 则为112.00 ng。如果使用荧光定量PCR方法定量, 最低基因组DNA用量可降为46.00 ng。
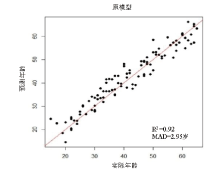
100份浙江汉族男性血斑样本作为验证集, 对已建立的9-CpG年龄推断模型[8]进行年龄验证, 其验证结果见图2。对于100份男性样本来说, 总体MAD值为2.95岁。按照年龄段分为5个组(A-E组), 分别为16~25、26~35、36~45、46~55、56~65岁, 分别统计MAD最小为2.70岁, 最大为3.21岁, 对于前4组(16~55岁), ME均为正值, 第5组(56~65岁)为负值(见补充材料表S1)。从图2可以看出, 每组高估与低估的样本个数基本相同。
96份浙江汉族女性血斑样本在9个CpG位点测得的甲基化值采用已建立的9-CpG年龄推断模型[8]进行年龄计算, 并与实际年龄进行对比, 总体MAD值为3.18岁(见图3)。从补充材料表S2可以看出A— D组的个体推断年龄均为高估, 且每个组都只有个别样本推断年龄比实际年龄小; E组的预测年龄基本为低估, 且ME值与前面四组比较, 差异具有统计学意义(P=1.0×10-3)。为明确男性及女性样本在模型验证中的差异, 对男性及女性样本各个位点的甲基化值进行t检验, 结果显示CpG6、CpG7位点有显著性差异(见补充材料表S3)。为进一步优化模型, 在96份样本中选取70%的样本作为训练集, 30%的样本作为验证集, 使用R软件“lm”函数对原模型进行调整优化, 优化后的模型为:
y=13.757+11.521×CpG1– 18.614×CpG2– 19.604×CpG3– 14.395×CpG4+24.885×
CpG5+7.945×CpG6+21.563×CpG7– 29.438×CpG8+87.226×CpG9
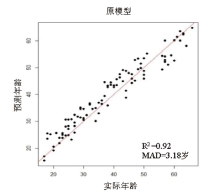 | 图3 年龄推断原模型在女性样本中实际年龄与预测年龄的散点图Fig.3 Scattering dot plot from actual age against the predicted with female samples by the 9-CpG age prediction model |
训练集和验证集的R2分别为0.94和0.93, MAD值分别为2.59岁和2.78岁(图4)。对新模型与原模型的所有样本预测年龄与实际年龄差值的绝对值进行配对t检验, 结果具有统计学差异(P=0.03), 所有样本采用新模型进行年龄推断计算所得总体MAD为2.65岁, 其预测误差值比原模型小0.53岁, 新模型的预测准确性更高。
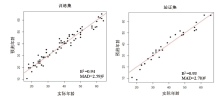 | 图4 新建立的年龄推断模型中实际年龄与预测年龄的散点图Fig.4 Scattering dot plot from actual age against the predicted with the modified 9-CpG age prediction model |
在新模型中, 96份女性样本在上述的分组中, 各组之间的MAD值较为均一, 与原预测模型相比, 其预测准确度均有提升, 且通过配对t检验, 显示MAD值在两种预测模型中存在显著性差异(见表2)。
| 表2 两种预测模型MAD的比较 Table 2 Comparison of MAD between two prediction approaches |
本文采用EpiTYPER技术平台对浙江汉族个体的血斑样本进行供者年龄推断研究, 验证以前建立的多元线性回归模型的检材适应性与适用人群范围。基于DNA甲基化的年龄推断在法医学领域的研究已有很多报道, 在不同的研究中, CpG位点的选择与组合不尽相同, 而且采用的预测模型也各有不同。有学者专门比较了基于血液样本的6种预测模型, 发现不同的预测模型, 其预测准确度存在差异。多元线性回归模型可最大程度保留原始数据, 且应用最为广泛, 适于低样本量数据建模[11]。EpiTYPER技术平台也是DNA甲基化研究中定量检测常用的方法, 通过交叉验证比较发现其所获得的数据准确性及重复性均较高[8]。
血斑DNA样本的甲基化检测具有挑战性。Suchiman等认为采用EpiTYPER技术平台检测DNA甲基化时, 要想获得较好的检测结果, 提取高纯度及高分子量的基因组DNA非常重要, OD值比(A260/A280)应在1.7~2.0之间[12]。由于附有纱布载体, 本文中血斑DNA的OD值比平均为1.69±0.16, 因此在进行PCR扩增时, 为了提高扩增效率, 增加了0.05U聚合酶以及引物浓度调整为2.0 μmol/L。重亚硫酸盐处理是甲基化检测非常重要的一步, 与常规血液样本DNA相比, 由于血斑提取到的DNA质量较差, 转化回收率对于后续检测更为重要。针对血液样本DNA来说, Sam等学者比较了6种不同的重亚硫酸盐转化商业试剂盒, 发现不同的试剂盒转化回收率差异较大[13], 但是对于血斑样本DNA尚无相关报道。本文采用EZ DNA MethylationTM转化试剂盒发现不同样本间的DNA转化回收率差异明显, 最高可达75.35%, 最低仅为11.05%。
本研究中100份浙江汉族男性血斑样本推断年龄MAD值为2.95岁, 与基于血液样本建立的北方地区男性个体年龄的预测准确性(MAD=2.89岁)基本一致, 而且不同年龄组间的MAD值无显著性差异, 显示9-CpG年龄推断适用于浙江地域的男性血斑样本的年龄推断。前期建立的9-CpG年龄推断模型针对的是男性汉族个体, 本研究发现直接使用该模型计算女性汉族个体的年龄普遍存在高估, 因此性别会影响年龄推断结果。尽管有一些研究结果与本文不一致, 如:Bekaert通过4个年龄相关位点检测了105份男性样本和101份女性样本发现所建立的年龄推断模型无性别差异[14]; Correia Dias等通过对51份葡萄牙人血液样本(男性18份, 女性35份)的检验, 也未发现男女样本间的MAD值有明显差异[15]; 国内孙晓萌等通过男女样本各40例在年龄偏差上的对比, 同样显示无统计学差异[16], 但笔者分析认为可能与上述研究选取的样本量小、CpG位点选取差异有关。有学者通过Illumina 450K阵列进行全基因组甲基化测序, 发现男性和女性个体与年龄相关的CpG位点不同, 即使相同的CpG位点, 其相关系数也存在差异[17]。
本研究中发现CpG6、CpG7这两个位点的甲基化值在男性样本与女性样本间存在着显著性差异, 导致原模型不适用于女性样本。使用96份女性样本重建线性回归模型, 总体MAD下降了0.53岁, 且不同年龄组间的MAD值较为均衡, 最小为2.24岁, 最大为2.90岁。
本文采用分光光度计测定的单管PCR扩增的BS-DNA量最低为12.0 ng, 这个用量比LEE等[18]报道的采用SNaPshot技术进行甲基化DNA检测所需的BS-DNA量(> 5.0 ng)稍高。但如果是采用实时荧光定量PCR法进行测定, 其BS-DNA的量也能降为5.0 ng。分光光度计测定DNA质量不仅快速且不需任何实验试剂耗材, 在大批量样本的研究中具有优势, 但其准确度易受到RNA、蛋白质的影响。而实时荧光定量PCR仪在一线法医DNA实验室中的使用更为普遍, 其测量精度也较高, 但是必须使用专门的定量试剂盒, 定量成本较为昂贵。
本文在研究大批量样本中采用了分光光度计, 通过两种不同定量方法的比较, 能为一线DNA技术人员利用血斑样本进行DNA甲基化年龄推断提供参考数据与借鉴。有效提高DNA转化回收率将是我们下一步研究的内容, 期望可进一步拓展9-CpG年龄推断模型在现场血斑检材和样本中的应用。
与本文相关的补充数据见:http://www.xsjs-cifs.com/CN/abstract/abstract6981.shtml。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|