第一作者简介:王斌,男,江苏盐城人,学士,主检法医师,研究方向为法医物证学。E-mail: wangbin8712@aliyun.com
本文研究上海浦东地区汉族人群Y-STR基因座和Y-SNP单倍群的基因分型,比较上海浦东汉族与其他人群之间的遗传关系,分析浦东汉族人群遗传结构。采用YfilerTM Platinum PCR扩增试剂盒对500名浦东汉族无关个体的38个Y-STR基因座进行基因分型,计算基因频率、单倍型多样性等群体遗传学指标。基于38个Y-STR基因座,应用EA-Ypredictor软件进行单倍群预测,应用Pedigree Tagging System家系标记系统进行单倍群验证并定义,并与其他人群进行比较。浦东汉族500名无关个体观察到493种单倍型。单倍型多样性(HD)为0.999 943 9,匹配概率(HMP)为0.002 056,鉴别能力(DC)为0.986 0,独特单倍型比例(FUH)为0.972 0。基于多维尺度分析结果表明浦东汉族与江苏汉族(Rst=0.000 9)和浙江汉族(Rst=0.000 9)遗传关系最近。Y-SNP分析结果显示,浦东汉族人群主要单倍群为O单倍群,比例为82.60%,O1a-M119的比例为25.20%。EA-Ypredictor的主要单倍群预测准确率达97.60%。38个Y-STR基因座在浦东汉族人群中有较高的遗传多态性,可以满足法医学应用。Y-SNP和Y-STR联合应用实现了对浦东汉族人群遗传结构的精细解析。本研究获得的基础数据应可为群体遗传学和法医学研究提供数据参考。
Both the forensic and population genetics build on fundamental genetic data and consanguineous relevancy. Therefore, such a purpose-oriented exploration was here conducted with the Han-ethnic population in Shanghai Pudong area through investigation of those pertinent Y-STR loci and Y-SNP haplogroups plus comparison of its genetic relationship with the other populations, hence having the correlating genetic structures analyzed. With utilization of Yfiler™ Platinum PCR amplification kit, the selected 38 Y-STR loci were genotyped from 500 unrelated individuals of Han-ethnic population among Pudong area. Calculation was carried out into the received genotypic data to deliver the related population genetic indexes, e.g., the gene frequency and the haplotype diversity. Moreover, the EA-Ypredictor was used for haplogroup prediction about the obtained 38 Y-STR loci, with the Pedigree Tagging System being chosen for haplogroup validation and definition, thereby having the haplogroup frequency data compared with the other populations. There were 493 haplotypes observed from the 500 Pudong Han-ethnic individuals, showing the haplotype diversity (HD) value as 0.999 943 9, the matching probability (MP) value as 0.002 056, the discriminative power (DC) value as 0.986 0, and the fraction of unique haplotype (FUH) value as 0.972 0. Furthermore, MDS analysis demonstrated that the Pudong Han-ethnic population had the closest genetic affinity to its same racial group in both Jiangsu (Rst=0.000 9) and Zhejiang (Rst=0.000 9). Also, Y-SNP exploration suggested that the Pudong Han-ethnic population have its main haplogroup as the O haplogroup that accounts for 82.60% and the O1a-M119 for 25.20%, indicting the EA-Ypredictor reached an accuracy of 97.60% for main haplogroup prediction. In conclusion, 38 Y-STR loci were tested from the Pudong Han-ethnic population, attaining such a high genetic polymorphism that could meet the application of forensic genetics. Additionally, EA-Ypredictor’s Y-SNP probing into the Y-STR data realized a fine analysis about the genetic structures of Pudong Han-ethnic population. The basic data obtained here should be able to provide relevant reference for population genetics and forensic research.
Y染色体非重组区(non-recombining region, NRY)遗传标记具有父系遗传、单倍型等特点, 是研究人类进化、群体遗传学、法医遗传学的理想标记[1]。Y染色体短串联重复序列(Y-short tandem repeat, Y-STR)和单核苷酸多态性(Y-single nucleotide polymorphism, Y-SNP)是NRY区两种重要的遗传标记, 其中, 利用Y-SNP单倍群构建系统发育树进行单倍群划分是进行地理祖先和种群推断的主要手段之一[2]。Y-SNP单倍群在不同地区和人群中的分布均表现出一定的特征性, 根据未知来源男性样本的Y-SNP单倍群, 可推断其地理来源或所属的群体[3]; 也可以利用高分辨率Y-SNP单倍群结合Y-STR单倍型进行家系溯源, 起到辅助司法案件侦破的作用, 故具有重要的法医学意义[4]。
上海浦东地区是其东南沿海的一座半岛, 为黄浦江东部地区的统称。据2020年人口普查结果, 浦东地区人口已达568万余。本研究应用Yfiler™ Platinum PCR扩增试剂盒[5](美国Thermo Fisher Scientific公司)对上海浦东地区汉族的38个Y-STR基因座(DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS437, DYS438, DYS439, DYS444, DYS447, DYS448, DYS449, DYS456, DYS458, DYS460, DYS481, DYS518, DYS522, DYS533, DYS549, DYS557, DYS570, DYS576, DYS593, DYS596, DYS627, DYS635, DYS643, DYS645, YGATAH4, DYS385a/b, DYS527a/b, DYS387S1a/b)进行基因分型, 采用Y-SNP家系标记试剂盒(中国阅微基因公司)[4]分析24个Y-SNP 位点, 定义11个主要单倍群和13个亚单倍群, 主要单倍群包括E-M96、D-JST021355、N-M231、C-M130、O-P186、I-M170、IJ-M429、K-M9、QR-M45、G-M201、IJK-M522; 亚单倍群细枝包括D1a1a1-N1、D1a2a-P47、O1a-M119、O1b-M268、O1b2-M176、O2-M122、O2a1-KL1、O2a2-P201、O2a2b-P164、O2a2a1a2-M7、O2a2b1a1- M117、C2-M217、N1a1-M46。Y-STR和Y-SNP联合分析, 实现了对上海浦东汉族人群遗传多态性的深度解析, 提高了鉴别能力, 扩大了遗传信息含量, 能够满足不同案件需要, 以及为群体遗传学研究提供基础数据支持。
采集上海市浦东地区500名汉族男性无关个体的血液样本并存留于FTA血样采集卡上(英国Whatman公司)。本研究所有男性无关个体均为至少三代以上世居的本地人, 基于知情同意原则, 浦东地区的研究对象(n=500)均签署了知情同意书。本研究所有内容均由复旦大学伦理委员会批准通过(批准编号为BE1806)。
使用打孔器将DNA血卡样本剪切成1 mm2大小, 采用Chelex-100法提取基因组DNA。使用Yfiler™ Platinum PCR扩增试剂盒分析所选38个Y-STR基因座。采用Y-SNP家系标记试剂盒对24个Y-SNP位点进行基因分型。扩增实验分别按照试剂盒说明书完成。使用9700型PCR仪(美国Applied Biosystems公司)获得扩增产物。PCR扩增产物以3500xL基因分析仪(美国Applied Biosystems公司)进行毛细管电泳分离。分型结果使用软件GeneMapperTM ID-X(美国Applied Biosystems公司)分析。
直接计算浦东汉族人群38个Y染色体STR基因座的等位基因数量、等位基因频率。采用R Studio软件(R语言版本号4.1.2, https://www.rstudio.com)计算法医学参数:单倍型匹配概率(haplotype match probability, HMP)、鉴别能力(discrimination capacity, DC)、独特单倍型比例(fraction of unique haplotype, FUH), 单倍型多样性(haplotype diversity, HD)。有关数据与其他群体共有的27个Y-STR基因座进行比较, 使用Arlequin软件计算群体间成对遗传距离Rst。
使用R Studio软件中的“MASS”软件包进行多维尺度分析。应用EA-Ypredictor软件[6]对38个Y-STR基因座进行单倍群预测, 采用ARMS-PCR验证预测的准确性。直接计算浦东汉族人群单倍群频率分布结果并与其他参考人群进行比较。
本研究Y-STR基因分型体系包括32个单拷贝基因座和3个多拷贝基因座(DYS385a/b、DYS527a/b、DYS387S1a/b)。单倍型数据见补充材料表S1, 经直接计算得到的基因频率的结果见补充材料表S2。
浦东汉族人群32个单拷贝Y-STR基因座共检测出240个“等位”基因, 基因频率分布为0.002 0~0.958 0。在多拷贝基因座DYS385a/b、DYS527a/b、DYS387S1a/b分别检测出62、37、46种单倍型, 单倍型频率分布在0.002 0~0.160 0之间。经计算, 38个基因座在浦东汉族人群中的单倍型多样性(HD)为0.999 943 9, 匹配概率(HMP)为0.002 056, 鉴别能力(DC)为0.986 0, 独特单倍型比例为(FUH)0.972 0。在DYS458、DYS448、DYS627、DYS518、DYS385a/b基因座中观察到有中间基因分型的现象, 在多拷贝基因座DYS527a/b、DYS385a/b、DYS387S1a/b则有多基因分型模式存在(中间基因分型和多基因分型均经重复三次实验验证)。38个Y-STR基因座的GD值计算结果见表1, 其分布在0.081 1(DYS645)~0.952 3(DYS387S1a/b)之间。在浦东汉族人群中共检出493种单倍型, 其中, 486种单倍型为唯一分型, 7种单倍型检出2次。
| 表1 38个Y-STR基因座在浦东地区汉族人群中的基因多样性 (n=500) Table 1 Genetic diversity of Y-STR loci from Chinese Han-ethnic population in Shanghai Pudong area (n=500) |
本研究Y-SNP基因分型体系包括24个Y-SNP标记。单倍群结果见表2。浦东汉族的主要单倍群结果为:C单倍群6.80%、D单倍群1.60%、N单倍群6.80%、O单倍群82.60%、QR单倍群2.20%。详细单倍群结果为:C2-M217(6.80%)、D1a1a1-N1(1.60%)、N1a1-M46(2.00%)、N-M231(4.80%)、O1a-M119(25.2%)、O1b2-M176(0.20%)、O1b-M268(7.00%)、O2a1-KL1(21.40%)、O2a2a1a2-M7(2.00%)、O2a2b1a1-M117(12.20%)、O2a2b-P164(10.60%)、O2a2-P201(1.80%)、O2-M122(2.20%)、QR-M45(2.20%)。
| 表2 浦东地区500名汉族人群的Y-SNP单倍群 Table 2 Y-SNP haplogroups from 500 unrelated Han-ethinc individuals in Pudong area |
根据浦东汉族和其他25个参考人群的共有Y-STR基因座“等位”基因频率, 计算成对遗传距离Rst。基于成对遗传距离进行多维尺度英文分析, 结果见图1。北方汉族参考人群包括黑龙江[7]、陕西[8]、内蒙古、山东[9]、河南[7]、长春[10]、辽宁[11]等地汉族群体; 南方汉族参考人群包括上海[12]、浙江[13]、江苏[14]、江西[7]、安徽[15]、宁波、重庆[16]、云南[4]、广东[17]、深圳、贵州[18]、四川[19]、广西[7]等地汉族群体。少数民族参考人群选择宁夏回族[20]、贵州苗族[21]。外部人群(Out Group)选自日本[22, 23]和韩国[24]。未注明参考文献的人群数据来源于YHRD数据库[25]。
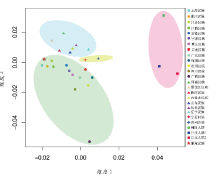 | 图1 浦东汉族和其他25个参考人群多维尺度分析Fig.1 Multidimensional scaling plot with Pudong Han-ethnic population and 25 reference ones |
北、南方汉族分别用三角形、圆形, 少数民族群体用四角星, 外部人群用方形, 而上海浦东汉族则用红色五角星表示。结果显示, 上海浦东汉族与江苏汉族(Rst=0.000 9)和浙江汉族(Rst=0.000 9)的距离最近。在中国人群中, 浦东汉族与广西汉族的距离(Rst=0.064 9)最远。
MDS图形整体呈现南北方人群分层, 北方汉族人群(蓝色圆圈内)聚类关系更加紧密, 南方汉族人群(绿色圆圈内)聚类较为松散, 这说明南方汉族人群相对于北方汉族人群在Y-STR基因座中有较大的遗传变异。在南方汉族人群内部, 浦东汉族、江苏汉族、江西汉族、上海汉族、浙江汉族更加接近。
基于38个Y-STR基因座的分型结果, 本研究使用EA-Ypredictor软件进行单倍群预测, 得到预测单倍群。应用Y-SNP家系标记试剂盒进行单倍群定义, 得到其真实单倍群结果。
经计算, 主要单倍群预测准确的样本为488个、不准确的12个, 准确率为97.60%。预测不准确的12例样本及其实际单倍群结果见表3。其他预测结果见补充材料表S1。
| 表3 12例预测错误样本及实际检验结果 Table 3 12 EA-Ypredictor-falsely-predicted samples ascribed into their erroneous and actual Y-SNP haplogroups |
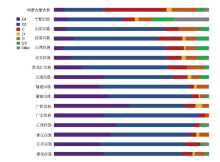
为了进一步探究中国人群的单倍群分布的相关性和差异, 基于已发表的论文数据[7, 26, 27, 28, 29], 本研究调查了其他15个中国人群主要单倍群的频率, 数据比较结果见图2。
本研究对浦东汉族人群的38个Y-STR基因座的遗传多态性调查结果显示, 浦东汉族HD值为 0.999 943 9, 表明该38个Y-STR基因座联合应用可满足对浦东汉族人群鉴别的需要。在38个Y-STR基因座中, 有4个基因座的GD值低于0.5, 分别是DYS391(0.412 2)、DYS437(0.445 9)、DYS438(0.377 3)、DYS645(0.081 1), 其他基因座的GD值均大于0.5, 这说明本研究所用的Y-STR基因分型体系有良好的遗传多态性, 适用于司法实践个体识别和亲缘鉴定。
基于遗传距离Rst, 本研究选择了其他25个人群进行多维尺度分析, 聚类结果显示出不同地域人群之间的亲缘关系。从整体上看, 26个人群可被划分为四类:南方汉族(包括上海、浙江、江西、江苏、安徽、宁波、重庆、云南、广东、广西、深圳、贵州、四川等地所选汉族), 北方汉族(包括黑龙江、陕西、内蒙古、山东、河南、长春、辽宁等地所选汉族), 少数民族(贵州苗族和宁夏回族)以及外部人群(日本和韩国人群)。东南地区汉族(包括浙江、上海、江西、江苏、浦东等地汉族)处于南北方汉族之间, 但是更接近南方省份汉族。既往研究曾引入一个“中部汉族”的概念来定义东南中部地区汉族人群[30], 本研究结果也表明南方省份汉族的东南地区群体具有更紧密的遗传关系, 这也同时证明南北方人群有其内部遗传结构[31]。
Yin等[32]对公开发表的Y染色体单倍群预测软件进行了比较, 在主要单倍群的预测中, EA-Ypredictor的平均准确率更高。基于对云南昭通人群的详细单倍群预测结果, EA-Ypredictor的表现更加突出, 详细单倍群预测准确率达89.50%。本研究中EA-Ypredictor的主要单倍群预测准确度达97.6%。未来投入到实际应用中可快速地将主要单倍群区分, 然后再进行精细单倍群结构的鉴定, 以降低实验成本, 缩短实验周期, 提高鉴定效率。Y-STR单倍型数据库在法医学实战中发挥着重要的作用, Y-STR作为男性特征性标识服从单倍遗传的特性并积累了相当多的突变, Y-STR的平均突变率在3.35×10-3突变/遗传标记/代[33], 这样高突变率的特征使得Y-STR在不同人群之间可能存在完全相同或相似的现象。2014年, Ballantyne等[34]收集了全球111个群体的1.4万男性无关个体数据, 基于17个Y-STR基因座, 发现了地理位置不同的群体之间存在相同单倍型的情况。Wang等[35]比较了2万例男性单倍型数据, 揭示出不同的Y-SNP单倍群的Y-STR单倍型存在相同或相似的情况。Y-SNP的突变率约为3×10-8突变/遗传标记/代 [33]。Y-STR的分化程度远高于Y-SNP, Y-STR的高突变率是不同单倍群之间存在相同或相似Y-STR单倍型的原因之一。在实际案件中, 如果目标Y-STR单倍型在数据库中匹配到多个家系时, 可以通过Y-SNP单倍群信息进一步缩小家系范围。也就是说, Y-SNP单倍群信息在Y-STR的基础上能够增加一个维度进行个体识别。
以往的研究表明, 中国汉族人群超过70%都是O单倍群[36]。在本研究中浦东汉族的O单倍群频率是82.60%, 和中国人群整体单倍群分布频率一致。O单倍群可以继续被划分为两个大支, 主要分为O1和O2单倍群, 这两个单倍群占东亚男性的60%。中国汉族人群人口众多, 单倍群频率数据可以反映出不同地理位置人群的遗传差异。O1单倍群的分布受地理格局影响, 其在北方省份低频分布, 几乎都在20%以下; 而在南方省份汉族人群中比较高频, 都在25%以上。本研究显示浦东汉族O1单倍群占比32.40%。其中O1a-M119占比25.20%, 是浦东汉族人群比例最高的单倍群。O1a-M119在中国东南沿海、侗傣族人群、台湾原住民集中分布[37]。O1a-M119单倍群在人群中差别比较显著, 虽然山东省和上海浦东地区都是中国内陆的半岛, 但是山东省O1a-M119的比例仅为3.0%[4]。以往的研究对O2单倍群下游的O2a2b-P164和O2a1-KL1的研究结果表明, O2a2b-P164在南方汉族的平均分布比例为21.54%[37], 在北方汉族分布则为34.11%, 故O2a2b-P164能够反映南北方人群的差异。而本研究中浦东汉族的O2a2b-P164和O2a2b1a1-M117单倍群的是22.8%, 这个频率分布的结果更符合南方汉族的特点。通过人群中Y-SNP频率分布, 可以推断未知个体的生物地理来源, 因而在面临Y-STR单倍型比对无结果的案件时, Y-SNP数据或可提供有效线索。
综上所述, 上海浦东地区汉族人群38个Y-STR基因座有较高的遗传多态性。Y-STR和Y-SNP的联合分析, 更精确地解析了浦东汉族人群的遗传结构, 可为司法实践的家系溯源和个体识别提供理论依据和数据支持。本研究所获得的Y-STR基因频率和Y-SNP单倍群频率数据还可为法医学与群体遗传学研究提供原始数据。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|