第一作者简介:余仲昊,男,河南驻马店人,硕士研究生,研究方向为法医病理学。E-mail: 1259970467@qq.com
目的 针对水体中常见的硅藻、蓝藻和气单胞菌,设计相应引物,采用qPCR方法进行检测,验证该方法的特异性和检测灵敏度,并探讨其在溺死诊断中的应用价值。方法设计与溺死相关的浮游生物特异引物ND、UPA、CBR、AER、HLYA、rPOD;扩增20种浮游生物标准株DNA以确定其特异性和灵敏度;检测38只实验兔的肺、肝、肾组织的样本,计算设计的6对引物扩增产物的总体检测阳性率,并与相应的硅藻电镜检验结果对比,验证所建立方法诊断溺死的有效性。结果 ND、UPA两对引物能特异扩增硅藻属,CBR能特异扩增蓝藻属,AER、HLYA、rPOD能特异扩增气单胞菌属;以上6对引物扩增产物均能达到0.0001ng的检测灵敏度。实验兔的qPCR检测结果显示,16只溺死兔肺组织均为阳性,且分别有13例肝和14例肾呈阳性,总体诊断阳性14例,阳性率可达87.50%;16只死后浸泡兔和6只空白对照兔中,只有5例死后浸泡兔的肺组织检出阳性,其余皆为阴性。电镜结果显示,16只溺死兔的肺、肝、肾均为阳性,16只死后浸泡兔和6只空白对照兔中,只有5例死后浸泡兔的肺组织电镜检出硅藻阳性,其余皆为阴性,与qPCR结果相一致;经统计学分析,上述两种方法对38只实验兔的总体诊断结果差异没有统计学意义( P=0.49>0.05),对38只实验兔的所有肺、肝、肾样本检测的诊断阳性率差异没有统计学意义( P=0.29>0.05)。结论 上述6对引物结合qPCR检测方法进行溺死诊断,具有较高的灵敏度,且特异性较好,具有良好的应用前景。
Objective To design the relevant primers and have qPCR adopted for detection of the related diatoms, cyanobacteria and aeromonas (those widely-distributed aquicolous microbes) and verified of the specificity and sensitivity about their forensic applicability into drowning diagnosis.Methods Six pairs of specific primers of UPA, ND, CBR, AER, HLYA and rPOD were designed for detection of drowning-related planktons (the three kinds indicated above). 20 standard strains of plankton were amplified of their DNA to determine the specificity and sensitivity. 38 rabbits (16 ones as the drowned, DG; 16 ones as the postmortem submersion, PSG; and 6 ones as the blank control, BCG) were tested of their tissues of lung, liver and kidney. The overall positive rate of the 6 pairs of primers was calculated through their amplified products and compared with the observation under electron microscopy so as to confirm the effectiveness of the established method for diagnosis of drowning.ResultsBoth ND and UPA had certain diatoms specifically amplified, so had CBR to cyanobacteria, and AER, HLYA, rPOD to aeromonas, respectively. The sensitivity of the 6 pairs of primers was 0.0001ng except for UPA’s 0.001ng with nitzschia. 38 experimental rabbits showed their qPCR results, leaving the 16 drowned ones being positive from all the lungs yet only 13 livers and 14 kidneys (rendering an overall positive rate as 87.50%); the 16 PSG ones being positive from 5 lungs while all their livers and kidneys negative; and the 6 BCG ones being all negative. Electron microscopy exposed that the 16 drowned rabbits were all positive with every lung, liver and/or kidney, being almost consistent with the qPCR results; the 16 PSG ones were positive from just 5 lungs and negative for all livers and kidneys along with the rest of lungs; and all samples of the 6 BCG ones were negative, too. Statistical analysis demonstrated no significance among the detection results of the 38 experimental rabbits between the two methods (the here-established qPCR detection and electron microscopy) ( P=0.49>0.05) and neither among the detection results of all the 38 rabbits’ lungs, livers and kidneys ( P=0.29>0.05).Conclusions The selected 6 primers are of high sensitivity and specificity for diagnosis of drowning when combined with qPCR detection, revealing good application prospect.
溺死, 是由于溺液阻塞呼吸道及肺泡, 阻碍气体交换, 造成体内缺氧及二氧化碳潴留而发生的窒息性死亡。水中尸体的死因诊断是法医工作的重点和难点。早期法医工作者常通过相关尸体征象, 如口、鼻部蕈样泡沫, 水性肺气肿等[1]来初步推断水中尸体死因是否为溺死。然而, 随着尸体在水中浸泡时间的延长, 尸体征象常因腐败而消失, 且一些其他死因的尸体也偶有上述征象出现, 故单独依靠尸体征象来诊断溺死的准确性较低。
硅藻检验诊断溺死有其历史, 法医工作者在光学显微镜下观察计算尸体脏器组织中的硅藻形态和数量而推断是否溺死。该法虽操作简单, 但由于光镜观察的局限性, 检验阳性率较低, 且其操作过程也较易造成污染[2, 3, 4, 5]。随着技术的进步, 通过对传统光镜下硅藻检验的改良优化, Zhao等[6, 7, 8]建立了微波消解-真空抽滤-自动扫描电镜法(MD-VF-Auto SEM), 以自动扫描电镜代替了传统的光镜, 既提高了检测灵敏度, 又使方法具有定量能力而将准确性大幅提高。但该法所需要的仪器设备价格较昂贵, 使用条件要求高, 难以在基层实验室广泛开展。
近年来, 随着分子生物学技术的不断发展, 已有不少研究者将DNA检测应用到溺死诊断领域中。以水体中的硅藻、蓝藻、气单胞菌等溺死相关浮游微生物的特定基因[9, 10, 11, 12, 13]为靶点, PCR毛细管电泳检测法(PCR-CE)可完成水中尸体脏器硅藻、气单胞菌等微生物的检测, 从而可诊断溺死。如麦柏盛等[14]采用PCR-CE法检测嗜水气单胞菌gyrB和16S rRNA基因, 彭帆等[15]检测rbcL基因, 均表明PCR-CE检测技术在一般案件中具有较高的实用价值。然而, 该法耗时较长、灵敏度也不高。
在PCR基础上发展起来的实时荧光定量PCR(real-time quantitative PCR, qPCR)是一种实时监控核酸扩增的技术, 通过在PCR反应体系中加入荧光基团, 利用荧光信号的变化对PCR过程进行实时监控, 以此实现对初始模板的定量分析。凭借其特异性强、灵敏度高和检测周期较短等特点, qPCR技术现在被广泛应用于临床病原检测、肿瘤诊断以及基因差异表达中[16, 17, 18]。在水质研究领域, 也有学者采用qPCR检测技术来监测水体中有害藻华的动态变化[19, 20]。
在我们前期研究发现并检测的有限硅藻种类中, 以硅藻UPA基因为靶点的qPCR检测技术在溺死诊断研究中具有较好的应用价值[21]。基于对广东省内珠江水体微生物的研究[22, 23], 针对水体中常见的硅藻、蓝藻、气单胞菌等浮游生物, 我们设计了6对特异性引物ND、UPA、CBR、AER、HLYA、rPOD, 拟构建一种qPCR溺死诊断方法, 以便能够更加全面、快速、灵敏地检测水体微生物的DNA, 从而为溺死诊断提供更多的证据信息, 为法医学溺死诊断再提供一种鉴定手段。
PowerSoilTM DNA Isolation Kit试剂盒、20 mg/mL蛋白酶K (Qiagen), DTT、Qubit® dsDNA HS Assay Kit DNA定量试剂盒 (Thermo fisher Scientific), NuHi® Robustic SYBR Green Mix(苏州新海), 浓硝酸(分析纯, 广州化学试剂厂)。
高速冷冻离心机(Sigma), Thermomixer Vortex振荡仪及恒温混匀仪(Eppendorf), Qubit® fluoro-meter v2.0荧光定量仪(Invitrogen), Milli-Q超纯水系统(Millipore), 高压蒸汽灭菌锅(Hirayama), QuantStudioTM 7 Flex Real-Time PCR System实时荧光定量PCR仪(Thermo Fisher Scientific), MW 3000微波消解仪(Anton Paar), HL-6型多联滤膜富集装置(珠海黑马仪器公司), FEI Quanta 600型扫描电镜(FEI公司)。
1.2.1 藻种、菌种标准株及人基因组DNA
根据前期研究基础, 针对广东省珠江流域水体中常见的溺死相关浮游生物种类, 特购置以下藻种、菌种标准株:4种淡水硅藻(小环藻Cyclotella sp.、针杆藻Synedra radians、菱形藻Nitzschia sp.、舟形藻Navicula sp.), 5种海水硅藻(三角褐指藻Phaeodactylum tricornutum、旋链角毛藻Chaetoisceros curvisetus、威氏海链藻Thalassiosira weissflogii、中肋骨条藻Skeletonema costatum、隐秘小环藻Cyclotella cryptita), 4种淡水蓝藻(念珠藻Nostoc sp.、鱼腥藻Anabaena sp.、产毒铜绿微囊藻Toxic microcystis、不产毒铜绿微囊藻Microcystis aeruginosa), 2种海水蓝藻(钝顶螺旋藻Spirulina platensis、聚球藻Synechococcus), 以上藻种标准株均购自厦门谱蓝生物科技公司; 5种水生气单胞菌(杀鲑气单胞菌Aeromonas salmonicida、嗜水气单胞菌Aeromonas hydrophila、舒氏气单胞菌Aeromonas schubertii、维氏气单胞菌Aeromonas veronii、温和气单胞菌Aeromonas sobria), 3种人体共生菌(长双歧杆菌Bifidobacterium longum、大肠杆菌Escherichia coli、白色念珠菌Candida albicans), 以上菌种均购自广东省微生物研究所; 人类女性基因组DNA 9947A购自Thermo Fisher Scientific公司。
1.2.2 实验兔样本
经南方医科大学实验动物伦理委员会批准(批准号:L2020064)后, 选取38只普通级新西兰兔作为实验对象, 雌雄参半, 体重在2 ~ 2.5 kg之间, 由南方医科大学实验动物管理中心提供。
实验兔随机分为甲、乙、丙三组:甲组为溺死组(drowned group, DG, n = 16), 将实验兔置于兔笼沉入水下溺死, 死后在水下浸泡30 min; 乙组为死后浸泡组(postmortem submersion group, PSG, n=16), 在远离水的地面上采用空气栓塞法将兔子处死, 待确认兔子完全死亡后, 将兔子置于水下浸泡30 min; 丙组为空白对照组(blank control group, BCG, n=6), 在远离水的环境中采用空气栓塞法将兔子处死, 后将兔子置于解剖台上等待解剖。为提高检出率及避免交叉污染, 每只实验兔均采用一次性器械解剖, 分别提取其所有的肺、肝、肾组织。同时收集溺死地水样2 L, -80 º C保存备用。
藻种和菌种培养液:取1 mL加入到含有细胞裂解液及研磨珠的PowerBread管中, 10 000 r/min离心30 min, 弃上清液0.5 mL。
器官组织样本:取0.5 g, 置于PowerBread管中, 采用灭菌后的剪刀剪碎, 然后于其中加入10 μ L的蛋白酶K和10 μ L的DTT; 恒温振荡仪56 º C振荡3 h, 振幅为1 500 r/min。
水样中浮游生物:取200 mL溺死地点水样, 经真空抽滤富集于滤膜上, 置于PowerBread管中, 采用灭菌后的剪刀将滤膜剪碎, 然后于其中加入10 μ L的蛋白酶K和10 μ L的DTT。
以上三种样本均采用PowerSoilTM DNA Isolation Kit试剂盒按说明书提取各自DNA。以Thermo Fisher Scientific公司Qubit荧光定量试剂盒制作标准曲线:1 μ L样品DNA, 199 μ L工作液, 振荡离心, 室温避光孵育3 min, 制作标准曲线并测定DNA浓度。
根据前期硅藻形态学研究基础, 针对广东省珠江流域内常见的硅藻、蓝藻及气单胞菌等浮游生物, 选取其在GenBank数据库中的6个特异性基因, 使用引物设计软件Primer Express 3.0.1设计特异性引物, 经过NCBI中Primer-BLAST的引物特异性验证, 最终筛选出6对高度特异的引物:ND、UPA、CBR、AER、rPOD、HLYA(表1)。委托上海生物工程技术服务有限公司合成这些引物。
| 表1 筛选所得的引物详细信息 Table 1 The selected primers and their detailed information |
25 μ L的qPCR扩增检测体系:NuHi® Robustic SYBR Green Mix 12.5 μ L, 正、反向引物各0.5 μ L(浓度均为 10 μ mol/L), DNA模板1 μ L, 扩增水10.5 μ L。qPCR扩增程序参数:95 º C × 5 min; 95 º C × 5 s, 60 º C × 25 s, 40个循环; 熔解曲线分析:60 º C × 1 min, 95 º C × 1 min, 升温过程中收集信号。使用QuantStudioTM 7实时荧光定量PCR仪进行扩增检测, 相关操作按该设备操作说明书进行。
分别使用上述6对引物, 扩增9种硅藻、6种蓝藻、5种水生气单胞菌以及3种人体共生菌和人类基因组DNA 9947A, 采用qPCR检测方法, 验证引物特异性。
经Qubit荧光定量仪检测所提取的藻种和菌种标准株DNA浓度后调整至1 ng/μ L, 续以10× 倍比浓度梯度稀释6次, 用qPCR方法确定上述6对引物对各种模板DNA的最低检测范围以确定引物的灵敏度。
使用上述6对引物, 经qPCR法检测实验兔的脏器组织; 采用微波消解-真空抽滤-自动电镜扫描法(MD-VF-Auto SEM)对实验兔的脏器组织及水样进行硅藻形态学检验。参照法医学诊断溺死的阳性标准, 以肺组织检出阳性为基础, 肝、肾任一组织检出阳性, 则诊断为溺死。
计算上述6对引物产物的总体检测阳性率, 与MD-VF-Auto SEM法检测结果阳性率相比较, 分析两种方法对溺死诊断的有效性。
采用SPSS 20.0做统计分析, 根据数据分布类型, 选择Fisher精确性检验比较上述6对引物分别对38只实验兔的溺死诊断正确率和38只实验兔的所有肺、肝、肾样本的溺死诊断阳性率是否与MD-VF-Auto SEM法具有显著性差异。检验水准α = 0.05, 若P < 0.05, 则差异具有统计学意义。
经qPCR, 用ND、UPA、CBR、AER、HLYA、rPOD等6对引物分别扩增上述的硅藻、蓝藻、气单胞菌、人体共生菌以及人基因组DNA。结果显示:ND引物能特异扩增舟形藻、三角褐指藻和旋链角毛藻; UPA引物能特异扩增小环藻、针杆藻、菱形藻、威氏海链藻、中肋骨条藻和隐秘小环藻; CBR能特异扩增念珠藻、鱼腥藻、产毒铜绿微囊藻、不产毒铜绿微囊藻、钝顶螺旋藻和聚球藻; AER能特异扩增杀鲑气单胞菌; HLYA能特异扩增嗜水气单胞菌和舒氏气单胞菌; rPOD能特异扩增维氏气单胞菌和温和气单胞菌。
6对引物均不能扩增长双歧杆菌、大肠杆菌和白色念珠菌等3种人体共生菌的DNA以及人基因组DNA 9947A, 表明上述引物特异性较好, 在尸体溺死诊断中不会受到宿主因素的影响。
Qubit测定标准株DNA浓度并调整至1 ng/μ L及10× 倍比浓度梯度稀释6次后, qPCR方法测定上述6对引物对各种模板DNA的最低检测范围而确定引物的灵敏度为:除UPA对菱形藻的检测下限为0.001 ng外, 其余引物对其特异扩增的藻种或菌种均能达到0.000 1 ng的检测下限, 表明该溺死诊断方法灵敏度较高, 能够在一定程度上解决因尸体腐败而造成的部分检材降解的诊断难题。
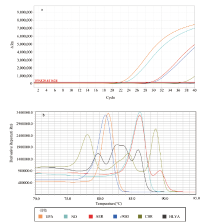
利用上述6对引物, 经qPCR方法分别检测了38只实验兔(16只溺死兔, 16只死后入水兔, 6只空白对照实验兔)的肺、肝、肾组织及溺死所用水样, 结果表明, 16只溺死实验兔的肺、肝、肾组织和溺死水样皆有不同程度的检出(表2); 在16只死后入水实验兔和6例空白对照实验兔的肺、肝、肾组织检测中, 只有5例死后入水兔的肺组织检测结果呈阳性, 检出引物分别是ND、UPA、AER和rPOD(如图1所示), 同时这5例肺组织的扫描电镜观察到的硅藻大多是小环藻和菱形藻等硅藻属(如图2所示), 表明在硅藻检测方面, MD-VF-Auto SEM和qPCR两种方法具有一定程度的一致性。
| 表2 兔子样本浮游生物检出率 Table 2 The detection rate of plankton from the handling-different sampling rabbits killed |
qPCR和MD-VF-Auto SEM两种方法对38只实验兔的诊断正确的结果以及对38只兔子的所有肺、肝、肾组织的诊断阳性分析的结果分别见表3, 经Fisher精确性检验分析, 上述两种方法对该38只实验兔的总体检测诊断结果差异不具有统计学意义(P=0.49> 0.05), 对38只实验兔的所有肺、肝、肾样本检测的结果诊断阳性率差异也无统计学显著性(P=0.29> 0.05)。
| 表3 实验兔诊断结果汇总分析表 Table 3 Summary about the diagnosis results from the experimental rabbits |
这表明在溺死诊断方面, 本文所建立的qPCR方法和MD-VF-Auto SEM法一样, 具有较高的准确性, 具有应用于实际检案中的潜能。
溺死研究, 不少学者采用多种技术、多层次分析诊断, 比如形态学层次的硅藻电镜检验方法, 分子生物学层次的藻类16S rRNA、SSU以及水生气单胞菌areA等基因靶点的PCR-CE检测方法。
基于以上研究, 本文选择珠江水体中常见的浮游生物, 包括硅藻、蓝藻、水生气单胞菌等, 针对不同物种的不同基因, 设计特异性引物(ND、UPA、CBR、AER、HLYA、rPOD)。
经过特异性实验验证, 引物ND、UPA能够特异性扩增小环藻、针杆藻、舟形藻、菱形藻等硅藻属; 引物CBR能够特异扩增念珠藻、聚球藻、鱼腥藻等蓝藻属; 而针对水中常见的水生气单胞菌如杀鲑气单胞菌、嗜水气单胞菌以及维氏气单胞菌等, 引物AER、HLYA和rPOD则能分别完成特异性扩增。而用上述6对引物对常见的人体共生菌(长双歧杆菌、大肠杆菌、白色念珠菌)以及人类基因组DNA 9947A扩增检测均呈阴性, 故正常条件下, 上述引物在实际案件检测中不会受到人体宿主DNA及体内常见共生菌的干扰, 具有较高的特异性。
同时, 引物灵敏度实验表明, 除引物UPA对菱形藻的检测下限为0.001 ng外, 其余引物对其特异扩增的藻种或菌种均能达到0.000 1 ng的检测下限, 扩增产物熔解曲线平稳, 峰尖且窄, 表明该6对引物灵敏度较高, 能够在一定程度上解决因尸体腐败而造成的部分检材降解的诊断难题。
应用qPCR方法检测已知的溺死/非溺死实验动物尸体中的水生浮游生物(硅藻、蓝藻、气单胞菌属)DNA分子标记, 在16例已知溺死动物尸体中有14例检出水生浮游生物, 剩余2例只在肺中检出而肝、肾未检出, 根据硅藻检验结果诊断溺死原则, 该2例检出结果不能判定为阳性。
此外, 在16例死后浸泡以及6例空白对照的实验兔肺、肝、肾组织检验中, 有5例死后浸泡实验兔的肺组织检出阳性, 而肝、肾则为阴性, 表明这5例的检出结果与实验设计相一致, 均为死后浸泡, 即非溺死。此5例的肺组织检验呈阳性, 可能是它们在水中浸泡时, 水中浮游生物经呼吸道进入到肺组织所致。
上述结果表明以qPCR法为基础的浮游生物DNA检测方法具有可行性, 对溺死案件的诊断能起帮助作用。
按照溺死标准, 本研究中16例溺死实验兔的肺、肝、肾组织以及溺死所用水样经MD-VF-Auto SEM法检验浮游生物, 结果阳性率均为100%, 而qPCR检测方法对肺、肝、肾以及溺死所用水样的浮游生物检出率分别为100%、81.25%、87.50%和100%, 总检出率为87.50%; 16例死后浸泡兔的检测, MD-VF-Auto SEM法和qPCR法都只在5例肺组织中检出阳性:扫描电镜下观察主要为小环藻和菱形藻等硅藻属, 但数量不多, 计算总数分别为36个/10 g肺组织和41个/10 g肺组织; qPCR的检出阳性引物分别为UPA、ND、AER和rPOD, 硅藻检出结果与扫描电镜检出结果相一致。因扫描电镜观察细菌的前处理操作较为复杂, 且对实验环境以及仪器的要求更严格, 本次实验未进行细菌层面下两种方法的比较。
MD-VF-Auto SEM方法检材用量较多(肺3 g、肝10 g 和肾10 g), 过程耗时, 且易产生污染实验环境的酸雾或污染物, 对实验设备和操作技术要求也高, 故难以同时处理大量样本。qPCR法检材用量少(肺、肝和肾各 0.5 g), 从DNA提取到qPCR结束仅需4~6 h, 分析扩增曲线即可得到结果, 无须电泳检测扩增产物的步骤, 操作简便、省时, 又不产生环境污染物, 因而有利于处理大量样本。且在检测全面性上, 本研究建立的qPCR检测方法能同时检测硅藻属、蓝藻属以及气单胞菌属。因此, 在大规模快速筛查检测方面, qPCR法更具优势。
但是需要指出的是, 在诊断准确率方面中, qPCR法稍低于MD-VF-Auto SEM法, 原因可能是:
1)检材提取因素:溺死过程中, 浮游生物进入各器官、组织后并不是均匀分布, qPCR法的单次检材提取量较少(肺、肝、肾均0.5 g), 取材覆盖面较低, 可能无法包含组织中所有的浮游生物, 从而导致诊断准确率低于检材提取量多(肺3 g、肝10 g、肾10 g)的电镜法。
2)浮游生物活性因素:电镜法为藻类形态学检验, 观察到的是藻类坚硬的外壳, 即使藻类活性丧失, 其外壳仍不受影响, 电镜下仍可检出阳性。而qPCR法为核酸检验, 浮游生物的活性丧失、提取方法不够成熟导致的DNA缺失等问题就会直接影响到后续的DNA模板质量, 从而导致该法的诊断准确率较电镜法略低。
综上, 本研究基于硅藻、蓝藻和气单胞菌检测所建立的qPCR溺死诊断方法, 虽存在诊断准确率稍低的问题, 但其具有所需检材少、检测周期较短、检测通量大等优点, 随qPCR检测和DNA提取技术的不断完善, 将其与MD-VF-Auto SEM法联用, 将能极大提高硅藻以及其他浮游生物在溺亡者内脏中的检出率, 降低电镜法所致的假阳性率, 增强溺死相关浮游生物检验诊断溺死的证据力。尽管现在还缺少qPCR诊断溺死的标准, 但该法作为溺死诊断的辅助性手段, 对于溺死样本的快速筛查检验相关技术研发与拓展应会有较好的应用前景。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|