第一作者简介:李甫,男,天津人,学士,副主任法医师,研究方向为法医物证学。E-mail: 13911779804@163.com
随着DNA技术的发展和应用拓广,案件现场生物检材及其提取检验愈受重视、愈显重要性,近年来混合检材的DNA有效检出比例越来越高,混合STR分型的拆分与合理解释也益发成为DNA鉴定技术领域的研究热点与难点。本文主要针对当下国际主流的两款混合分型分析软件——STRmix和云算GPM进行比较研究,就两者的基本信息、理论、分析过程作比较,并结合实战应用和典型案例综合分析两者的异同,希冀为法医DNA工作者提供选用参考和使用依据,也希望能够推动国内混合STR分型分析的发展及与国际交流互鉴。
The ever-advancing DNA technology has been bringing the on-site biological evidential materials into more increasing attentions together with their collection and DNA testing, resulting in successful more detections of eligible STRs from mixed DNA evidential substances in recent years. Accordingly, more profiles of mixed STR are being encountered in forensic casework and waited for reasonal deconvolution and appropriate interpretation in routine task of DNA laboratory, leaving them becoming a hotspot and one of the greatest challenges for forensic DNA genotyping. STRmix and cloud-calculating GPM are presently the main-current two pieces of software to interpret mixed STRs of DNA, the most commonly used software worldwide, too. Discussions were here carried out on the two pieces of software about their similarities, differences, interpretation-dependent theories, analysis process and actual applications plus cases for exampling. References are intended to provide forensic DNA peers with the elucidated interpretation of mixed STR profiles and promotion of international exchange and cooperation.
现场勘查质量的提高, DNA检验技术日新月异的发展, 仪器检测灵敏度的增强和实验技术人员操作水平的提升, 都使得越来越多原来无法利用的现场检材DNA可应用于案件分析与侦破工作, 如低模板量DNA以及混合DNA[1]。在实际案件中, 混合STR分型分析可以为侦查提供线索, 为诉讼提供证据, 能够大大提高混合生物样本的利用价值。但混合STR分析对技术人员的专业基础及经验要求较高, 同时还需要熟练运用概率统计学等知识对结果进行科学合理的分析解释, 因此, 其又成为目前法医学工作者面临的难点与挑战[2-3]。
传统的人工拆分方式主要针对两个供体的混合样本, 多数情况下需利用其中已知供体的基因型, 结合峰高、峰面积等参数推算混合比例, 进而拆分出另外一个供体的基因型。这种方式虽针对性强, 但过于依赖个人经验和判断, 且没有统一量化标准, 其结果不能作为定案的唯一物证, 只能作为侦查线索或参考证据[4]。近几年, 随着技术的发展, 一种智能化混合STR分析方法应运而生, 即利用混合STR分型分析软件进行分析。该方法引入统计学概率模型, 能更充分地利用混合图谱中的数据信息, 同时设置可量化的标准, 重复性好, 减少了主观性, 拆分结果更加客观 [5, 6, 7]。目前, 国内混合STR分型分析主要依赖人工拆分方式, 分析软件的应用还极少。本文针对目前国际上已获广泛认可的两款混合分型分析软件STRmix和云算GPM, 从基本信息、理论比较、分析过程和实战应用等多个方面进行分析与比较, 以期为当前国内案件中混合STR分型分析这一难题提供解决方案, 为法医DNA工作者提供选择参考和依据, 也期望能够推动国内混合分型分析及与国际交流互鉴。
STRmix混合图谱分析系统由新西兰皇家科学院环境科学研究院(ESR)与南澳大利亚法庭科学研究中心(FSSA)共同设计开发, 严格遵循SWGDAM Interpretation Guidelines for Autosomal STR Typing by Forensic DNA Testing Laboratories 3.3.2所认证的概率型基因分型分析技术标准, 目前已被包括美国FBI、英国KFS在内的全球多家犯罪学实验室购买使用, 其对混合图谱的解释结果得到了广泛认可[8]。
云算GPM混合图谱分析系统其核心为德国MDG集团的全连续法概率分型软件GenoProof® Mixture 3, 国内瑞源公司对其进行二次开发, 针对软件的网络应用方面也进行优化。GenoProof® Mixture 3软件按照SWGDAM Interpretation Guidelines for Autosomal STR Typing by Forensic DNA Testing Laboratories 3.3.2要求进行验证, 同时也符合德国DNA委员会对混合斑解释建议要求, 分析结果可作为法庭示证使用[9]。
2.1.1 基本原理
混合分型分析软件按照技术原理划分, 主要有二进制法、半连续法和全连续法三类[10]。
1)二进制法通过设定阈值来处理峰的随机性并筛选数据, 该方法舍弃了一部分数据信息, 在处理低拷贝DNA、降解DNA、混合比例差异较大DNA及三人以上混合DNA时结果不理想。
2)半连续法优于二进制法, 但忽略了峰值的变异性、混合比例及stutter峰所占百分比等因素, 在拆分的合理性、准确性上还有待提高[11]。
3)全连续法建立了较为完善的生物模型, 综合了影响STR峰值信号的全部因素, 并且联合MCMC先进算法, 模拟分析图谱中包含的所有信息, 最终以概率的形式给出可能的分型组合[12, 13], 是目前公认最先进的混合图谱拆分系统。
本文讨论的两种混合分型分析软件STRmix和云算GPM均属于全连续法分析系统, 其主要计算原理是通过建立生物模型来计算每个等位基因的总产物量[14], 通过MCMC算法的迭代运算不断优化计算其中的各个参数, 直到找到能够解释证据图谱的最佳基因型组合。
2.1.2 结果呈现形式
目前对于混合分型图谱的解释, 常见以下四种方法:定性评价、计算匹配概率、计算排除概率、计算似然率(likelihood ratio, LR), 国际法庭遗传学会推荐采用似然率法进行分析和解释[15, 16]。STRmix和云算GPM均可以结合参照样本的基因型进行对比综合分析计算, 并以似然率的方式呈现在结果报告中。
STRmix和云算GPM在模型构建时除了考虑等位基因峰高和峰面积之外, 还充分考虑到低拷贝数目、等位基因丢失、随机插入、杂合子不均衡等情况。其中云算GPM还可根据需要进行二进制法LR计算、全连续法LR计算、随机个体不被排除概率(RMNE)、随机匹配概率(RMP)等。
2.2.1 数据处理
两款软件数据导入形式差异较大。STRmix仅支持经GeneMapper或GeneMarker分型软件分析完毕的数据所导出的文本文件(或csv格式文件), 且对生成的文本文件有格式要求, 需在上述数据分析软件中作预设。云算GPM集成了更加完整的全流程数据分析功能, 支持导入HID或FSA格式的原始数据, 可以在软件内部进行数据分析, 手动分析完毕后也支持样本文件的生成, 进而进行下游的计算工作。
2.2.2 自定义功能
两款软件在自定义功能的内容和数量上存在着不同点, 具体差异见表1。
| 表1 软件自定义功能对比 Table 1 Comparison of application-defined functions between two pieces of software |
2.2.3 报告输出
STRmix的计算结果会以弹出的Calculation Results界面预览展示, 分析完毕的结果数据会根据类型保存在本地指定目录的不同文件夹内, 根据需要可以分别打开并查看。生成报告时, 支持生成PDF文件或Codis格式文件, 并通过勾选去掉不需要输出至报告的内容。
云算GPM的Results界面列举各位点基于当前假设的似然率、检(辩)方假设概率以及总似然率, 不同信息分列在不同的下拉选项卡中, 客户端分析的结果数据会上传至服务器中存储。生成报告时, 可选择PDF或Word形式, 并通过勾选去掉不需要输出至报告的内容。
混合DNA样本由两名或两名以上个体的血液、精液、分泌物、排泄物、脱落上皮细胞等同种或不同种类型物质附着在不同的犯罪现场载体上而形成。
一类是由不同个体的同一种体液或组织混合而成, 常见如多个个体的混合血痕, 多人受伤的伤害、杀人案件中提取的检材。
另一类是由不同个体的不同体液或组织混合而成, 最常见为性犯罪案件中提取到的检材, 主要为阴道擦拭物、被害人内裤、床单、卫生纸、犯罪嫌疑人阴茎拭子等, 这类检材包含精子和阴道上皮细胞, 属男、女个体成分的混合物。混合STR分型的分析对还原现场重建、案情调查均有帮助, 还能提供嫌疑人线索, 对协助案件侦破等有重要作用。
近年来混合检材的DNA有效检出比例越来越高, 部分混合样本可以通过实验技术分离组分后进行分析, 如激光捕获显微切割[17, 18]、差异裂解[19]、单细胞分离[20, 21], 但均有一定的难度和局限性。
使用STRmix、云算GPM等混合分型分析软件, 利用统计学的方法进行智能化计算, 可方便高效地进行多人混合拆分, 目前最多可拆分5人混合, 同时对拆分出来的基因型组合提供统计学权重。
经对2018— 2019年检验的案件进行梳理, 筛选来源均已知的2人混合分型图谱共54例(其中性犯罪类案件检材37例, 盗抢类案件检材6例, 伤害杀人类案件检材6例, 其他案件检材5例), 3人混合分型图谱1例(为故意伤害案件检材), 分别应用STRmix和云算GPM进行计算, 均拆分出了准确的供体分型。
另外, 选取盗窃类案件来源均未知的2人混合分型图谱25例, 3人混合分型图谱7例, 分别应用2种软件进行计算, 其结果报告按基因座分别以概率的形式给出了可能的分型组合, 再选取其中权重较高的分型通过全国公安机关DNA数据库进行快速比对, 并将比中的人员提供给侦查部门进行后期甄别。
简要案情:某日, 某地某商户发生一起盗窃案件, 店内监控显示嫌疑人曾翻动过收银台上物品, 勘查人员使用植绒棉签分别提取收银台上夹子、POS机、USB线插头、U盘拭子。
经检验, USB线插头拭子检出混合STR分型结果(图1), 其中可能包含有价值的破案信息。对该混合STR分型分别使用STRmix和云算GPM两种软件进行分析, 拆分其中主要贡献者。STRmix软件拆分结果报告见图2, 云算GPM的结果报告见图3。
 | 图1 USB线插头拭子分型结果Fig.1 The detected STR genotypes of DNA extraccted from the case-seized USB cable plug |
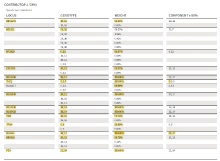 | 图2 STRmix分析结果报告主要贡献者部分Fig.2 The main contributor of Fig.1-indicated STR loci interpreted with STRmix software |
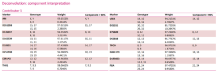 | 图3 云算GPM分析结果报告主要贡献者部分Fig.3 The main contributor of Fig.1-indicated STR loci interpreted with GPM software |
结果显示, 两个软件拆分出的主要贡献者在14个基因座上基因分型均一致且权重均大于90%, 在D21S11基因座两者分别给出了可能性较高的分型及对应的权重。
将上述拆分出的男性STR分型通过全国公安机关DNA数据库进行快速比对, 成功比中1名违法犯罪人员, 该信息为案件侦破提供了关键线索。
STRmix和云算GPM这两款软件的核心算法均基于目前最先进的全连续法分析系统, 通过统计学概率模型构建和MCMC算法进行综合分析, 以概率的形式给出可能的分型组合, 两者实际应用于混合图谱拆分过程时, 针对不同的计算需求存在一定的差异性。
STRmix的优势在于其每一步计算过程均提供高自由度的参数修改编辑功能, 可使有更多需求的使用者在充分考虑每一个变量的前提下, 在进行每一次的计算过程并获得更有针对性的分析结果的同时, 能尽量避免反复计算, 节约时间和人力成本。但该软件在正式应用于案件分析之前, 需要进行大量的内部验证实验, 以获得针对相关实验室的具体设置参数, 该过程较为费时费力。而云算GPM的内部验证工作主要由软件开发公司完成, 如果各实验室的操作流程规范、仪器、试剂、耗材等方面均符合软件的操作指南, 则可以使用通用参数设置得出符合其标准的运算结果。
云算GPM的优势在于融入了原始数据图谱分析功能, 可直接导入HID或FSA格式的原始数据并在软件内部进行数据分析的工作, 做到边分析边计算。在连接公安数据库后, 能对各个基因座的拆分结果按照概率进行排列组合, 将所得的大量可疑供体分型自动进行快速比对。与STRmix将各项参数的编辑修改功能整合至单项计算窗口中不同, 云算GPM拆分操作过程相对简单, 但其大部分参数设置和功能预置需要在分析前进入独立的设置菜单中修改, 在实时修订参数、实时计算数据方面整体流畅度不如STRmix, 有可能无法完全满足有特殊或复杂计算需求的使用者。
综上, STRmix和云算GPM两款软件作为当下国际认可度较高的混合STR分析工具, 均是基于统计学概率模型, 遵照权威标准进行计算, 其拆分数据结果有可量化的标准; 实际应用过程中, 两者在内部验证、参数修订、数据流转等方面有一定差异。目前, 国内混合STR分析主要依赖于传统的人工拆分方式, 分析软件的应用可以为解决案件中常见的混合STR分型分析这一难题提供参考和依据, 但具体的软件选择还应结合实际应用需求确定[3]。
法医DNA领域现在正经历着复杂分型的解释方法学转变。与现有的二元法相比, 概率统计学分析软件为解释微量、复杂的混合STR分型提供了有力的理论依据[22]。然而目前这些分析软件不能也不应该替代经验丰富的技术人员[10, 23]。对STR图谱仍需先进行标准分析和严格评估, 在充分理解软件计算原理的条件下, 完成数据的输入(混合DNA分型)直至输出(似然率), 将拆分软件作为一种帮助工具而非判定依据。恰如Buyckleton等所言:“ 为了理解分型比对软件的优势和劣势, 使用者和相关获益者最好能理解系统基本原理, 并以最全面的、深思熟虑的方式使用该系统以实现公共利益” [24]。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|


