第一作者简介:刘海,男,江苏启东人,硕士,副主任法医师,研究方向为DNA鉴定。E-mail: 13838287856@163.com
目的 探查中国河南汉族个体与年龄相关的DNA甲基化位点,构建年龄推断模型,进行甲基化和年龄相关性分析。方法 采用焦磷酸测序法对ELOVL2、ClOrf132、KLF14、TRIM59和FHL2基因的34个CG位点进行甲基化分析,利用SPSS 23软件的多元回归方法建立模型,对甲基化和年龄相关性做分析。结果 除ClOrf132基因3个CG位点的甲基化水平与年龄呈负相关外,其余4个基因的31个CG位点甲基化水平均与年龄呈正相关。多元回归分析表明,年龄与CG位点的甲基化水平存在明显的线性关系,实际年龄与推断年龄偏差在5岁以内的准确度达80%以上。结论 本研究构建的河南汉族个体年龄推断模型,有助于通过检测血液等组织的DNA甲基化水平推断个体的年龄范围,具有法医学应用前景。
Objective To survey the age-related DNA methylation sites in Chinese Han-ethnic population so as to construct an age inference model for analyzing the correlation between DNA methylation and age.Methods 34 CpG loci of ELOVL2, ClOrf132, KLF14, TRIM59 and FHL2 genes were analyzed into their DNA methylation through pyro-sequencing. The resulted data were processed with SPSS23 multiple regression approach so that a relevant model was established for correlative analysis of DNA methylation and age.Results The methylation level of 31 CpG loci of four genes (ELOVL2, KLF14, TRIM59 and FHL2) was of positive correlation with age, with the other three CpG loci of ClOrf132 gene showing negative for the methylation-age correlation. Multiple regression analysis revealed that there was a linear relationship between age and methylation level, demonstrating an accuracy of more than 80% within deviation of 5 years old between actual age and the inferred.Conclusion The age inference model here-constructed of China’s Henan Han-ethnic population is able to help determine the age range of individuals by way of detecting the DNA methylation level of tissues (e.g., the blood) such that the application prospects could be expected in forensic medicine.
DNA甲基化(DNA methylation, DNAm)是指DNA在甲基化转移酶的作用下, 以S-腺苷-甲硫氨酸为甲基供体, 形成5-甲基胞嘧啶的化学修饰过程[1]。DNA甲基化发生于1.5%的基因组相关DNA中, 在个体发育过程中DNA甲基化水平呈明显降低趋势[2]。DNA甲基化一般发生在CG位点, 人类基因组中除部分CpG岛之外的CG位点一般都趋向甲基化。对于转录活性基因, 启动子区CpG岛的胞嘧啶一般处于未甲基化状态, 而基因内部和重复序列中CpG岛的胞嘧啶维持高甲基化状态, 这样就维持了启动子基因组的稳定性。DNA甲基化是一种重要的表观遗传学修饰形式, 可调控基因表达、胚胎发育和癌症的发生发展[3, 4]。近年来的研究还发现, DNA甲基化与衰老密切相关, 基因组总体DNA甲基化水平随年龄增加而降低, 但部分位点的甲基化水平却随年龄增加而升高, 特定位点的DNA甲基化还与年龄呈线性相关[5]。如肿瘤相关基因hTERT的DNA甲基化与年龄呈负相关, ELOVL2基因的DNA甲基化水平可作为年龄预测的关键因子, 若有其他因子协同共预测还会使预测准确性进一步提高[6, 7]。因此, 本研究通过检测基因CG位点甲基化水平来寻找与年龄相关的CG位点, 进而通过DNA甲基化水平来进行年龄推断。
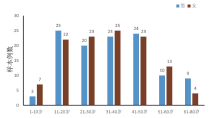
选取郑州申友医学检验实验室的剩余外周血样本231份, 采样经郑州第十六医院伦理委员会同意并由供者签署了知情同意书。取样年龄范围在1~80岁之间, 其中男性114例, 女性117例, 中位年龄36岁。为保证年龄推断模型的适用性, 本实验尽量对每个年龄阶段样本都选取, 并尽可能保持每个年龄段样本数量大致相同。不同年龄阶段人数见图1。
所有样本以人淋巴细胞分离液收集单核细胞并以生理盐水洗涤, 然后根据QIAamp DNA Blood Mini Kit(凯杰生物, 德国)试剂盒说明书提取DNA。取1 μ g的样品DNA, 采用DNA甲基化转化试剂EpiTect Fast DNA Bisulfite Kit(凯杰生物)按说明操作, 将DNA序列中非甲基化的C转变为U, 而甲基化的C保持不变, 以用于后续DNA甲基化水平检测。
根据文献[8]选取ELOVL2、ClOrf132、KLF14、TRIM59和FHL2基因作为年龄相关甲基化检测基因, 利用凯杰甲基化引物设计软件Assay Design 2.0设计筛选此5个基因甲基化水平检测引物, 其中一条引物采用生物素标记以验证特异性。引物由英维捷基(上海)贸易有限公司合成, 具体信息见表1。
| 表1 5个基因引物及甲基化信息 Table 1 Primers and methylation information relating to the selected five genes |
以50~100 ng经亚硫酸盐处理的DNA为模板, 使用甲基化专用PCR扩增试剂盒PyroMark PCR Kit(凯杰生物)进行25 μ L体系扩增, 程序为95 ℃、15 min; 94 ℃、30 s, 56 ℃、30 s, 72 ℃、20 s, 45个循环; 72℃、10 min。
| 续表1 |
按凯杰公司操作说明书将PCR扩增产物和有关试剂进行测序反应, 即:将2 μ L链酶亲和素加入到PCR产物中使其充分结合, 制备成单链, 然后加入测序引物和退火缓冲液, 85 ℃变性2 min, 冷却至室温使引物与样本退火杂交, 以备上机使用。按PyroMark Q24操作说明建立assay, 加入相应的PyroMark Gold Q24 Reagents测序试剂进行测序, PyroMark软件用于分析测序结果。
将231个样本分成两组, 一组由151个样本建立年龄训练集, 一组以80个样本建立测试集, 样本均为随机分配。采用IBM SPSS Statistics 23的多元线性回归方法建立年龄推断数学模型。多元线性回归可同时分析所有基因上的甲基化位点与年龄的相关程度, 同时按照各基因甲基化的情况筛选出R2大于0.9的模型公式。MAD (mean absolute deviation)用来表示真实年龄和推断年龄的平均绝对偏差, MAD的值越小, 表明模型推断年龄的准确度越高。
根据Zbieć -Piekarska等[8]的研究结果, 以P< 0.05为选择条件, 选取ELOVL2、ClOrf132、KLF14、TRIM59和FHL2基因共34个CG位点在中国河南人群中进行甲基化检测, 结果表明ClOrf132基因的3个CG位点平均甲基化水平较高, 甲基化率分布范围分别为51%~97%、8%~92%和28%~93%。而KLF14基因的4个CG位点平均甲基化水平较低, 甲基化率最低值为1%而最高值也都在50%以下。ELOVL2、TRIM59和FHL2基因所有检测CG位点的平均甲基化水平均处于中等水平。各CG位点甲基化水平见表1。
将训练集151个样本每个CG位点的甲基化水平都与其关联年龄进行相关性分析, 结果表明ClOrf132基因的3个甲基化位点与年龄呈负相关, 而其他四个基因的所有CG位点甲基化水平与年龄均呈正相关。
与年龄相关程度最高的甲基化位点为ELOVL2基因的E1位点和FHL2基因的F1位点, 甲基化相关系数为0.9, 呈现出高相关性, 而KLF14基因的4个甲基化位点与年龄的相关性最低, 均低于0.55, 其中KLF14基因的K4位点的值更低至0.2, TRIM59基因的8个甲基化位点与年龄的相关系数处于0.6~0.8之间, 属于几个基因的中间值。34个甲基化位点与年龄的相关系数具体值见表1, 80个样本测试集的甲基化位点与年龄的相关系数呈现出与训练集相似的结果。
为寻找更好的年龄推断模型, 使用IBM SPSS Statistics 23软件的多元线性回归方程方法对151个样本训练集的所有甲基化位点进行计算, 按标准化后R2大于0.9的设定得到以6个基因甲基化位点进行年龄预测值评估的数学模型公式:
M1=29.748+0.46× F1-0.528× C1+0.288× E4+0.291× T4
M2=29.184+0.383× F1-0.514× C1+0.205× E4+0.289× T4+0.293× E2
M3=32.887+0.446× F1-0.53× C1+0.19× E4+0.294× T4+0.315× E4-0.118× F9
M4=29.766+0.178× F1-0.524× C1+0.198× E4+0.276× T4-0.119× F9+0.286× F2
M5=29.616-10.531× C1+0.216× E4+0.277× T4+0.378× E2-0.11× F9+0.411× F2
M6=29.328-0.524× C1+0.288× E4+0.183× T4+0.327× E2-0.128× F9+0.378× F2+0.195× T5
注:上述等式中M代表模型公式
其中F、C、E和T分别代表基因的首字母, 数字代表基因的甲基化位点位置, KLF14基因由于各位点与年龄相关系数较低, 建立模型时被剔除。
采用6组年龄模型计算各自的MAD值, 分别计算3~7岁每岁的年龄预测准确度, 5个模型公式年龄推测模型的MAD值达到了4岁以内, 而模型6的MAD值为5.23 , 故排除该年龄模型。以MAD值在5岁以内为标准预测年龄准确度, 模型4的预测准确度最高为82%(R2为0.93), 模型6最低, 仅为57%, 其余均在70%以上。因此, 本研究选取模型4作为预测模型。
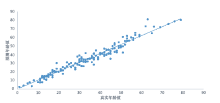
采用模型4, 对不同年龄分组的人群进行年龄推断, 图2为151例样本实际年龄和推断年龄的散点图。随后, 再将151个样本分为5组, 分别为1~18、19~40、41~50、51~60岁及60岁以上, 仍以5岁以内为年龄偏差上限, 结果发现, 1~18岁组准确性最高达到了93.94%, 随着年龄增加, 准确度逐渐降低, 各组年龄(由低到高)预测准确性分别为93.94%、85.71%、83.87%、64.29%、44.44%。将1~18岁组再分成为中童(1~10岁)、大童(11~14岁)和青少年(15~18岁)3组, 中童、大童和青少年组的标本数分别为7、13、14。采用模型4对年龄进行预测, 3组预测准确度分别为100%、90.91%、93.33%, MAD值分别为2.24、2.43、1.93岁, 大童和青少年组无显著性差异, 虽然青少年组的预测准确度最低, 但其MAD值却最小, 可能与样本量较少有关, 也可能是儿童、青少年发育阶段和成年人之间存在更深层次的生物学差异。
DNA甲基化总量在胚胎时最高, 随着年龄增长逐渐降低。因此, DNA甲基化可作为年龄预测分子标记, 在个体发育和细胞分化、诊断癌症等年龄相关疾病以及法医学确定年龄范围等方面有广泛应用[9, 10]。但迄今大多数研究对象均为欧美人群, 汉族人群较少[11, 12, 13]。本研究基于焦磷酸测序技术平台, 根据Zbieć -Piekarska等[8]的文献选取5个甲基化基因在231例汉族个体中进行DNA甲基化水平检测。结果表明所检测基因DNA甲基化水平可分为高(ClOrf132)、中(ELOVL2、TRIM59和FHL2)及低(KLF14)三个水平, 不同基因甲基化水平有较大差异, 与Vidaki 等[14]的研究结果一致, 这一差异有可能是不同基因或不同CG位点的功能不同所致。
所有CG位点与年龄进行相关性分析, 结果表明有的基因甲基化水平与年龄呈正相关(如ELOVL2、KLF14等), 有的位点甲基化水平与年龄呈负相关(如ClOrf132), 这是由于不同基因对年龄预测的作用不同。此外, 同样是正相关的CG位点, 相关系数也差别较大, ELOVL2基因的E1位点和FHL2基因 的F1位点, 甲基化相关系数为0.9, 而KLF14的基因的K4位点仅为0.2。其次是预测模型, Silva等[13]的研究表明一些基因可以单独利用回归模型进行年龄推断。本研究根据相关系数高低剔除了KLF14基因, 预测软件根据其余4个基因甲基化水平和相关系数推荐了6个统计分析模型, 由于不同模型中加权系数不同, 所以每个模型的预测准确度不同, 通过对同一年龄样本采用不同模型预测, 结果表明模型4的预测准确度最低亦达90.91%, MAD值为2.24。Zbieć -Piekarska等[15]在2014年的研究报道以ELOVL2基因对124例2~75岁的人群进行年龄推断, 准确度为83%, MAD值为5.75; 第二年, 该团队又对包括ELOVL2基因在内的8个基因利用回归模型对427例19~75岁的人群进行年龄推断, 准确度为86.7%, MAD值为2.7[8]。与Zbieć -Piekarska的结果相比, 本研究模型4的准确度和MAD值都较好, 因此选择模型4作为最佳的年龄推断模型。由于本研究和Zbieć -Piekarska的研究所用甲基化位点一致, 研究方法也类似, 模型4对年龄推断的准确度和MAD值稍好, 再次证明了这几个基因的甲基化位点会有助于对河南人群的后续年龄推断, 两者间的差异可能是不同人群遗传背景不同所致, 也可能与样本量较少有关。
为了进一步分析年龄与预测准确度的关系, 将年龄再分组为1~18、19~40、41~50、51~60岁及60岁以上5组, 结果表明1~18岁组准确度为93.94%, 而60岁以上组仅为44.44%, 年龄越大预测准确度越低。继续将1~18岁再分组, 结果表明1~10岁预测准确性最高, 达到了100%, MAD值也较小, 随着年龄增大预测准确性降低, MAD值增大。这一结果与Zbiec-Piekarska[8]和李姗飞等[16]的结果一致, Zibiec-Piekarska利用8个基因的41个CG位点进行年龄评估, 发现随着年龄增长精确度下降, 反之亦然。李姗飞等利用8个CG位点的年龄预测模型, 发现随着年龄的增加, 预测准确性出现相应的下降, MAD值大体上也逐渐增大, 年轻组(小于25岁)MAD值为2.42岁, 而年长组(65~75岁)为3.87岁。推测造成这一差异的原因有可能是随着年龄的增长, 胚胎时期形成的甲基化模式受到的环境影响越来越大, 也有可能是年龄相关性疾病影响了甲基化对年龄的预测。
最近, 丰蕾等[17]报道了采用DNA甲基化推断年龄的成功案例, 为法医学中的实际应用。但目前基于DNA甲基化推断个体年龄的准确方法还较少。本研究得出了本地汉族人群中几个基因甲基化与年龄的相关性情况及相关系数, 筛选出可预测年龄的合适的甲基化位点及统计模型, 对以后通过检测血液等生物检材的甲基化水平推断个体的年龄范围应有所帮助, 将有助于缩小嫌疑人的排查范围, 具有很好的应用前景。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|