第一作者简介:赵一霞,女,山东诸城人,硕士,法医师,研究方向为法医遗传学。E-mail: 18146501798@163.com
同卵双生子具有相同的基因组DNA序列,因而无法使用常规的DNA分型方法如短串联重复序列即STR分析对其作鉴别区分。故破获涉及同卵双生子的案件有非常大的挑战性,因此在法医物证学领域迫切需要新的技术方法来应对并解决这一难题。随着表观遗传学的发展,转录组学成为法医学研究的新切入点。MicroRNA(miRNA)作为一类内源性的非编码小RNA分子,在机体中参与调节多种生理学过程,是转录组学研究的重要对象。研究表明,miRNA具有高保守性、分子量小、表达时序性及组织特异性强等特点,表现出法医物证应用方面的巨大潜力。本文综合分析了miRNA检测技术在同卵双生子甄别中的应用可行性,并综述了近年来miRNA在同卵双生子甄别上的研究进展。
It is extremely problematic to discriminate between monozygotic twins (MZTs) because they share same genomic DNA sequence, causing them difficult to identify through the traditional DNA-sequence-based analytic methods such as STR (short tandem repeat) profiling. Therefore, the cases involving MZTs are posing great challenges to forensic scientists. Thus, new technical methods are demanded about forensic biological evidence. With the progressing development of epigenetics, the transcriptome has been becoming a new attraction for forensic researchers. MicroRNAs (miRNAs), among the important epigenetic research subjects, are endogenous small non-coding-for-protein RNA molecules, participating into many physiological regulatory processes. Accordingly, owing to the characteristics of high invariability, low molecular weight, ubiquitous abundance, temporal correlation and strong tissue-specificity, miRNAs have been showing potential in forensic applications. This paper reviews the recent research progress of miRNAs for eligible identification of monozygotic twins into forensic genetics.
个体识别是物证鉴定的重要研究内容, 现已建立起多种基于DNA分型的鉴定方法, 如短串联重复序列(short tandem repeats, STR)和单核苷酸多态性(single nucleotide polymorphisms, SNP)就是该领域应用最广泛的技术。但对于涉及同卵双生子(monozygotic twins, MZT)的案件, 涉事主体来自同一个受精卵, 具有相同的基因组DNA序列, 因此无法运用当前的个体识别方法进行甄别。如何科学、准确地实现MZT鉴别区分, 是物证鉴定工作者需要攻克的难题。1942年奥地利发育生物学家Conrad Waddington首次提出“ 表观遗传学” 的概念, 即在基因的核苷酸序列不变的情况下, 基因表达呈现出的可遗传变化现象[1]。表观遗传修饰如DNA甲基化、组蛋白修饰、微小RNA(microRNA, miRNA)介导的基因表达调节所展示的对外部刺激的敏感性等, 被视为基因与环境之间的“ 桥梁” 。事实上, 当MZT在不同生活环境或发生疾病的状态下, 两个个体通常会出现不同程度的表型差异, 因此为运用表观遗传水平差异甄别MZT提供了理论依据和新技术突破口。
miRNA是一类长度约18~23核苷酸(nt)的内源性非编码蛋白的小RNA分子。近年来大量的研究发现, miRNA可在体液(斑)组织来源鉴定、个体年龄推断、机体死亡时间推断及MZT甄别等方面发挥重要作用。本文主要就miRNA在MZT甄别方面应用的机制、研究进展和检测方法等内容进行综述。
1993年, Lee等[2]首次在秀丽隐杆线虫(Caenorhabditis elegans, C.elegans)体内发现一个长度为22 nt的RNA小分子lin-4, 其能在翻译水平抑制核蛋白lin-14的表达, 从而参与调节线虫幼虫的发育过程。2000年, Reinhart等[3]在C. elegans体内发现另一个长度为21 nt的RNA小分子let-7, 其具有转录后调节功能, 调控C. elegans由第一幼虫阶段过渡到第二幼虫阶段。lin-4和let-7 RNA基因是非同源的, 但却均是一组蛋白质编码靶基因的3’ 非翻译区序列的互补成分, 这些靶基因通常受到RNA的负调节, 由于lin-4和let-7都具有控制发育阶段的功能, 因此也被称为时序性小RNA(small temporal RNAs, stRNA)。在随后不到一年的时间里, 研究人员在其他真核生物如果蝇、蠕虫和人体细胞当中发现了超过100个(其中果蝇中发现约20个, 蠕虫中约有60个, 人体细胞中约有30个)这类可转录的非编码小RNA分子, 并将其命名为miRNA[4, 5, 6]。Sanger研究所建立的miRBase数据库(http://www.mirbase.org/)是储存miRNA信息最主要的公共数据库之一, 可为研究者提供包括已发表的miRNA序列数据、注释、预测基因靶标等信息。目前该数据库已更新至最新版本V22.0, 包含有271个物种的38 589个成熟的miRNA序列, 其中人类基因组编码的miRNA前体达到1 881个, 成熟miRNA达2 588个。但该数据库用于miRNA信息查询较多, 靶关系预测还较少。
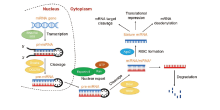
成熟的miRNA是一类长度约为21~25 nt的单链小分子, 通常5’ 端带有一个磷酸基团, 3’ 端为羟基端。3’ 端有1~2个碱基长度变化, 5’ 端第一个碱基对U有强烈的倾向性, 而对G有抗性。成熟的miRNA与上游或下游的序列不完全配对从而可形成茎环结构。从初级转录大分子RNA到成熟的单链miRNA至少需要三个步骤[7, 8]:1)miRNA基因由RNA聚合酶II/III转录生成初级转录产物pri-miRNA; 在微处理复合物Drosha-DGCR8的作用下, pri-miRNA被剪切成约70 bp、具有茎环结构的前体miRNA (pre-miRNA); 2)Ran-GTP依赖性核浆转运子Exportin5将剪切好的pre-miRNA转运至细胞质; 3)胞质中的pre-miRNA在RNase III Dicer的作用下被剪切成22 nt左右的二聚体RNA。该二聚体RNA(即miRNA/miRNA* )由miRNA以及长度与其相似、但不完全互补配对的茎-环结构的对侧臂 miRNA* 组成。二聚体RNA与含有Argonaute蛋白的装载复合体结合, 在解螺旋酶(helicase)作用下, miRNA* 从二聚体中解离并被降解, 成熟miRNA与复合体整合成RNA介导的沉默复合体(RNA-inducing silencing complex, RISC), 如图1所示。
随着越来越多的miRNA被发现, miRNA的命名及标准也经历了一些变化。最早发现的几个miRNA基因根据其功能被发现者分别命名为lin-4、let-7、lsy-6。2003年Ambros等[9]科学家联合发表一篇miRNA鉴定、注释及命名规则的文章。此后几年时间, Griffiths-Jones等[10, 11, 12]对miRNA的命名规则又进行了补充。当前, 以miRBase数据库为基础, 研究者建立了包括动植物、病毒miRNA前体及成熟体的完整命名体系。以动物miRNA为例, miRNA成熟体命名遵循如下几点:1)在确定命名规则之前发现的miRNA保留其原名字; 2)miRNA成熟体简写成miR, 根据其物种名称(hsa、mmu、rno分别代表人、小鼠、大鼠)及被发现的先后顺序加上阿拉伯数字, 如hsa-miR-122, 一般情况下, 数字越小代表发现越早; 3)高度同源的miRNA在数字后加一个英文小写字母(a、b、c)以示区别; 4)由不同染色体上的DNA序列转录加工而成的具有相同成熟体序列的miRNA, 则在后面加上阿拉伯数字以示区分, 如hsa-miR-199a-1/hsa-miR-199a-2; 5)通常一个miRNA前体长度约70~80 nt, 很可能两个臂分别产生了成熟miRNA, 在之前的规则中, 表达水平较高的miRNA尾部不加任何符号, 表达水平低的miRNA尾部加* 号。在miRBase19.0中, miR* 命名终止使用, 取而代之的是“ -5p” 和“ -3p” 命名, 代表从前体的5’ 端臂和3’ 端臂加工而来(如图2所示)。
大量研究表明, miRNA通过负调节信使RNA(messenger RNA, mRNA)广泛参与个体发育、细胞分化、增殖以及凋亡等生理过程和肿瘤的发生发展、心脏病、糖尿病等病理过程[13, 14]。其主要通过两条途径调节基因表达:一是miRNA介导RISC识别特异的靶mRNA的3’ 非翻译区, 然后直接剪切mRNA和/或抑制其编码蛋白的翻译, 从而抑制转录后基因水平的表达。RISC的作用方式取决于miRNA与mRNA互补程度, 若两者间互补配对程度高, 则RISC将对mRNA进行直接剪切; 若两者间互补配对程度不高, 则需通过抑制mRNA蛋白翻译过程。当mRNA降解后, miRNA保持完整并继续指导RISC对其他特异的靶mRNA发挥剪切作用。二是通过调节DNA甲基转移酶表达, 维持细胞中DNA甲基化或改变组蛋白修饰等多种途径而调节基因表达[15]。研究表明, miRNA既是DNA甲基化转移酶(DNA methylation transferase, DNMT)的效应器又是其调节靶标, miRNA结合DNA甲基化转移酶mRNA的3’ -UTR并抑制其表达, 从而影响全基因组的甲基化模式, 该机制在机体的许多疾病发生发展中发挥作用。
miRNA在法医学上的应用具有许多明显优点:1)与传统的血清学检测方法比较, miRNA的检测方法对检材需求量小, 灵敏度和特异性较高, 故能极大地减少假阳性的产生, 适用于犯罪现场的微量检材。2)与常用的mRNA分析(约200~300 nt)相比较, 其分子量小(约18~22 nt), 可稳定保存, 更加适用于法医案件中的降解或受损检材。3)具有表达时序性和组织特异性[16, 17, 18], 表现在生物发育的不同阶段、不同组织中均有不同的miRNA表达, 如C. elegans中的旁系同源基因和直系同源基因lin-4和let-7 RNAs在发育过程中具有阶段性表达。miR-1可特异性高表达于哺乳动物心脏, 而miR-122则在肝脏中具有表达特异性, miR-223主要在小鼠骨髓的粒细胞和巨噬细胞中表达。可以作为体液(斑)组织来源鉴定、个体年龄推断、机体死亡时间推断等分析的标记分子, 从而可为案件审判提供依据。
miRNA分析可成为法医学领域同卵双生子甄别新的突破口, 除基于上述生物学特征以外, 另一个更重要的原因在于, 同卵双生子中miRNA的表达是具有差异性的。虽然同卵双生子的DNA序列相同, 但在不同外部环境条件(饮食习惯、运动、健康状况等)影响下, 其个体的基因表观遗传修饰会出现差异, 最终表现为MZT的个体表型差异, 这就是使用miRNA分析鉴别同卵双生子的理论基础。Espin-Perez 等[19]的联合实验表明在低水平污染的空气中短时间暴露能够导致Hsa-miR-197-3p、Hsa-miR-29a-3p、Hsa-miR-15a-5p、Hsa-miR-16-5p和Hsa-miR-92a-3p发生显著变化, 可以作为指示空气污染的潜在标记分子, 并且可以作为肺癌等与空气污染相关的疾病的指示分子。在神经退行性疾病, 如帕金森和阿尔茨海默病[20]中, miR-151a-3p和miR-191-5p分别在这两种疾病中的表达具有显著变化。Zarrinpar等[21]对患有非酒精性脂肪肝炎(non-alcoholic fatty liver disease, NAFLD)的29对同卵双生子和11对异卵双生子血清中的miRNA进行检测, 其中在同卵双生子中鉴定到5个具有显著差异的miRNAs, 包括miR-30b、miR-140、miR-30c、miR-324-3p和miR-331-3p, 可以作为NAFLD的标记分子。Wu等[22]通过大规模平行测序技术(massively parallel signature sequencing, MPSS)对9对男性MZT外周血中的miRNA进行分析以筛选可标记人体寿命长短的环境相关miRNA。因此, 对遗留在犯罪现场的检材与相关的MZT两个体的miRNA表达谱作分析比对, 或将为确定嫌疑人身份提供一种可能。
随着MPSS技术的应用普及, 研究者尝试用全基因组测序的方法检测MZT之间存在的基因序列差异, 如拷贝数变异(copy number variation, CNV)、Y染色体上的DYZ1基因差异等, Bruder等[23]利用芯片技术检测了包含10对健康MZT在内的共29对MZT外周血中DNA的CNV, 结果显示在MZT个体中存在CNV。但从当前的情况来看, 全基因组测序技术非常耗时、昂贵且需要高质量的基因组DNA, 更由于MZT在基因组上的差异很小, 该方法区分MZT的有效性非常低。Wang等[24]运用STR分型技术鉴定到DNA序列上一个罕见的vWA基因座三带型嵌合突变, 从而成功地区分出了一对MZT, 但该突变的发生具有偶然性。已有研究表明, DNA甲基化的差异也可用于MZT的区分, Li等[25]对22对成年健康同卵双生子的血液样本DNA甲基化水平进行了全基因范围的检测, 筛选出了近百个差异明显的CpG位点, 但该方法也存在检测程序复杂, 对检材质量和数量要求都较高, 难以在法医学实验室中普及的不足。因此, 法医物证学领域仍需要尝试研究新的技术方法来解决这一难题。基于miRNA分析对于物证鉴定研究的独特优势以及进行MZT甄别理论和技术方法的可行性, 筛选可靠的miRNA标记分子进行MZT鉴别仍是新的研究方向。而近年来, 国内外也逐渐出现了相关的研究报道。方晨等[26]应用全基因组miRNA芯片技术对1对MZT外周血中miRNA的表达谱进行表达差异分析, 结果显示在MZT外周血中共检测到509个miRNA, 其中有96个在外周血中表达量较高且存在差异, 并从中筛选出3个血液中表达丰度较高的miR-151a-3p、miR-451a和miR-486-5p, 经利用qRT-PCR验证, 其结果与芯片结果相一致。但由于该研究仅使用1对MZT作为检测样本, 存在偶然性, 因此只可作为使用miRNA进行MZT鉴定的参考, 后续还需更多MZT样本的miRNA表达信息以进行详细的验证和分析。肖超等[27]同样使用miRNA芯片技术检测2对不同年龄、性别的MZT间miRNA的表达并对表达谱进行差异分析, 再采用qRT-PCR技术对分析得到的差异大的miRNA进行验证。结果表明男性MZT中共有74个差异表达的miRNA, 女性MZT中共有220个差异表达的miRNA, 筛选其中miR-92b、miR-520d-5p、miR-22* 和miR-338-3p共4个miRNA进行验证, 结果与芯片检测结果一致。Fang等[28]运用MPSS技术对4对健康汉族MZT(4个男性, 4个女性, 共8个个体)血液中的miRNA表达谱进行了分析, 结果显示, 平均每个个体中检测到158个miRNA, 在每对同卵双生子中有14%的miRNA表达具有差异性。再对表达水平差异较大的几种miRNA以qRT-PCR进行验证分析, 结果表明miR-451a在4对MZT中均表现出显著差异, 此外另有9个miRNA在2对MZT中表现出显著差异。这些结果为miRNA进行同卵双生子甄别鉴定提供了有力支持, 至少可作为重要的数据参考。
当前检测miRNA常用的技术方法有Northern印迹杂交法、基因芯片技术、实时荧光定量PCR技术等。近年来, 随着研究者对表观遗传学研究的深入, 转录组学成为物证鉴定研究中新的切入点, 转录组测序(RNA-seq)技术应运而生, 即运用高通量测序研究特定组织或细胞在某个时期转录出来的RNA表达量, 进而对相关基因和表型的关系进行分析, 常用的有基因芯片技术、大规模平行测序技术。Pritchard
等[29]对miRNA分析技术的基本细节以及各种生物样本中已建立和正在建立的miRNA检测技术有综述。对于寻找甄别同卵双生子的miRNA标记分子, 常用的策略通常是先采用转录组测序分析技术进行初步筛选, 并对获得的miRNA表达谱进行表达差异分析, 然后再使用实时荧光定量PCR技术对差异较大的目标miRNA进行准确的定量验证, 以求获得能够区分同卵双生子个体的特异性miRNA。
基因芯片(gene chip), 又称微阵列芯片技术(microarray)、生物芯片或DNA芯片技术, 该技术指在一块芯片上固定大量的与miRNA序列互补的探针, 然后经与标记的样本RNA杂交, 通过检测每个探针分子的杂交信号强度而获取样品分子的数量和序列信息。该方法能实现高通量研究miRNA表达, 短时间内同时检测大量miRNA的表达丰度, 是快速检测全基因组内特异性miRNA表达丰度的优先选择。但该方法对RNA的纯度质量要求较高, 在纯化过程会丢失掉许多小分子RNA, 影响低丰度RNA的检测, 虽然信息量大却质量低, 因此常伴有假阳性结果, 故通常用于前期目标分子的筛选[30]。
下一代测序(next generation sequencing, NGS)平台的出现为miRNA的检测提供了新方法。转录组测序技术(RNA sequencing, RNA-seq)是一种新兴的以基因测序为基础的高通量测序(High-throughput sequencing)方法。该方法首先从总RNA中分离纯化20~30 nt的miRNA, 分别在miRNA的5’ 端和3’ 端连上接头序列, 经逆转录得到70~80 bp的DNA片段, 制备成测序文库; 然后通过对文库中数以万计的克隆群组(colony)进行延伸反应, 检测对应的信号; 最终再对产生的阵列图像进行时序分析, 获取序列信息。该方法最主要的优势在于能够对已知和未知的miRNA进行检测, 并且可以精确读取miRNA序列信息。Goodwin等[31]曾对高通量测序技术原理以及各平台的优势和实践应用做过细致比较分析。最有代表性的平台有:Illumina的基因组分析仪、Roche454基因组测序仪以及AB Life Technologies的SOLiD系统。针对miRNA具有序列较短的特征, Illumina的基因组分析仪和SOLiD系统更为适用。其中, Illumina的基因组分析仪具有对样品需求量少, 数据误差小, 操作简便等优势。SOLiD系统测序通量大于20 GB/次, 读长可达50碱基(随机片段测序)和2× 50个碱基(末端配对测序), 单次运行所得的数据量最大、准确度最高。而Roche 454基因组测序仪读长为400~500 nt, 远大于miRNA的序列长度(22 nt), 因此该测序仪适用于对新物种的基因组测序和转录组测序, 极少用于miRNA测序。2013年, 该平台下GS FLX+和GS Junior的测序仪器因成本及应用范围限制等原因而停产。在短读长高通量测序平台中, 454平台和Ion Torrent平台的优势在于读长较长, 分别约为700和400 bp, 其中, Ion Torrent平台通量从50 Mb到15 Gb, 运行时间从2~7 h不等, 是目前测序平台中最快的一个, 在转录组与可变剪切鉴定中具有很大优势。在单分子高通量测序平台中, 最常用的是PacBio Biosciences的单分子实时测序法, 但该方法在miRNA测序中未得到广泛应用。
实时荧光定量PCR技术(Realtime PCR, qRT-PCR)具有高灵敏度的优势[32], 是目前实验室常用的RNA定量检测方法。该方法在反应体系中加入荧光基团, 通过荧光信号累积可实时检测整个PCR进程, 在扩增的指数期连续检测荧光信号强弱变化而能即时测定特异性产物的量, 从而可对起始模板进行定量分析。与mRNA相比, 成熟miRNA序列较短, 又没有poly(A)尾, 故不能按常规方法设计随机引物进行逆转录和PCR扩增。采用qRT-PCR技术对miRNA进行定量检测常用的方法是利用茎-环状引物反转录miRNA, 即stem-loop RT-qPCR检测法[33], 该方法需要在反转录引物中设计一段与miRNA互补的特异性序列和一段共有序列, 与靶miRNA退火反转录可得到一个较长的反转录扩增子(cDNA), 共有序列提供了一个通用引物结合位点, 接着通过一个与miRNA序列特异的引物和一个通用引物进行PCR扩增。该方法特异性高, 样品消耗量少, 能检测出低丰度表达的目标miRNA。
由于MZT具有完全相同的DNA序列, 因此常规法医学个体识别技术很难对其区分。基于DNA甲基化表达谱差异、X染色体失活、拷贝数变异等表观遗传水平的MZT鉴别研究正蓬勃发展。miRNA作为表观遗传学的重要研究内容, 通过负调节mRNA的翻译和/或直接剪切mRNA以及调节DNA甲基化和组蛋白修饰而广泛参与机体许多重要的生理、病理过程, 在表观遗传学方面发挥着重要作用。随着转录组学研究的深入以及RNA在干燥载体上具有较好稳定性的发现[34], 越来越多的研究证实RNA分析在法医学上具有很大的应用潜力。与mRNA分子相比, miRNA分子量小, 不易降解, 具有对案件检材需求量小、检测方便、成本低等优势, 更加适用于犯罪现场检材的快速检测, 尤其在面对高腐败和微量样本上具有突出优势, 因此愈益受到法医学工作者的青睐。
虽然当前利用特异性的miRNA进行体液斑迹组织来源推断、年龄推断、毒物分析、损伤和死因推断等多个领域的研究已获得快速进展, 但在MZT鉴定方面的相关报道却很少, 将miRNA分析技术应用到MZT鉴定面临一些挑战:1)机体中miRNA的表达连同DNA甲基化、组蛋白修饰与DNA序列上的STR、SNP位点最大的区别在于前者是一个动态的调节过程, 在后天的生长发育过程中能够随着外界环境的改变而出现变化, 继续遵循现有的RNA分析研究方法, 寻找具有普适性的某种或某一类区分MZT个体特异性的miRNA作为标记分子, 对研究者来说具有极大挑战, 因此亟待拓展新的研究思路。2)当前对MZT个体中miRNA的差异分析更多的是在疾病状态下的对比分析, 缺少对健康MZT个体的系统分析, 而对于犯罪嫌疑人而言, 其健康状况处于未知状态, 因此不具有普适性。3)现有研究报道中分析的MZT比较少, 其数据很可能不具有代表性。
在日后的研究中, 一方面, 要广泛借鉴已筛选的miRNA标记分子的研究报道, 如应用已筛选到的特异性miRNA作为某些疾病(如癌症、心血管疾病等)的标记分子, 为法医实战中通过检测不同健康状态下MZT个体miRNA标记分子的表达, 缩小目标miRNA分子检测范围、节约时间和成本、甄别MZT提供参考依据; 另一方面, 需在已有的研究基础上增加研究样本的数量和覆盖范围, 尤其是加强对不同环境条件或生活习惯影响所引致的MZT个体差异表达miRNA的研究, 确保实验数据的全面性和可靠性, 以期建立起全面的备选miRNA标记分子及引物库。近年来, 转录组测序分析技术、定量检测技术的进步以及生物信息学的综合应用, 将助力于运用分子生物学技术锁定嫌疑MZT, 若个体经转录组测序并与现场样品进行qPCR比对验证, 确定出犯罪嫌疑人身份, 就将是MZT甄别鉴定的新技术手段和成功案例。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|