第一作者简介:金鑫,男,河南开封人,硕士研究生,研究方向为法医人类学、法医遗传学。E-mail:16210700108@fudan.edu.cn
* 通讯作者简介:李士林,男,吉林长春人,博士,副教授,研究方向为法医人类学、法医遗传学。E-mail: lishilin@fudan.edu.cn目的 建立适宜的复合扩增体系,并在中国东北汉族人群中验证所选择的DIP-STR遗传标记,以推动DIP-STR遗传标记应用于实际办案。方法 参照Diana Hall等人设计的单遗传标记扩增体系,建立合适的复合扩增体系并以中国东北汉族人群进行验证,对等位基因频率、单倍型数、观察杂合度、有效基因型等相关参数数据进行统计和计算。结果 构建了合适的复合扩增体系。经对107个样本验证估计得出等位基因频率,所选择8个DIP-STR遗传标记具有较高的有效基因型概率(I值)。结论 8个DIP-STR遗传标记在中国东北汉族人群中具有应用价值。
Objective Establishing an appropriate composite amplification system of DIP-STR genetic markers to promote their application into practical cases through verifying them in the Han population of northeast China.Methods Based on the single-genetic marker amplification system designed by Diana Hall et al, a suitable composite amplification system of DIP-STR genetic markers was to build up and verify in the Han population of northeast China. Statistical analysis and calculation were carried out into the relevant data of allelic frequency, haplotype amount, observed heterozygosity and informative genotypes.Results The composite amplification system of DIP-STR genetic markers was successfully established. With the estimation of allelic frequencies through verification from 107 samples into the established composite amplification system, the selected 8 DIP-STR genetic markers had a high probability of informative genotype (I value).Conclusion The eight DIP-STR genetic markers and the involving composite amplification system have forensic applicability in the Han population of northeast China.
在法医学领域, 混合不平衡DNA的鉴定一直是个难题。常染色体STR是目前法医学应用较多且相当成熟的遗传标记[1, 2], 然而对于混合不平衡DNA检材, 常染色体STR受限很多, 因为通常主要贡献者的DNA会掩盖次要贡献者的基因分型。但这些次要者的DNA在很多情况下却正是所需要的基因分型, 概因犯罪嫌疑人往往是次要DNA的提供者。从PCR等常规手段扩增出的片段分析可知, 混合DNA中次要组分能允许的最低比例为10%[3, 4, 5, 6]。而要精确识别DNA上的等位基因, 则次要组分在总体中的比例不能低于20%[7, 8]。若DNA混合比例严重不平衡, 主要组分会在PCR扩增时“ 掩盖” 次要组分, 而少数得到扩增的次要组分又极易在毛细管电泳中受到背景杂音的影响, 使其无法被测序仪或人工识别, 从而导致个体鉴别失败[9]。Y-STR可以运用于男女混合不平衡DNA的鉴别, 但对于全女性的混合不平衡DNA就无法发挥作用 [10]。此外, 当混合DNA来自于同一家系的男性时, 由于Y-STR处于Y染色体的非重组区域, 那么Y-STR也将无法准确地将DNA定位到家系的某一具体成员上[11, 12, 13]。为解决上述问题, Castella等[14]提出了一种新型遗传标记即DIP-STR, 它依靠缺失/插入多态性片段(Deletion-insertion polymorphism, DIP)和STR。DIP-STR遗传标记区域包含两部分, 一部分是具有特异性序列插入(L-长等位基因)或缺失(S-短等位基因)的区域, 另一部分是与之连锁的具有高度多态性的STR区域[14, 15]。DIP-STR主要应用于对非平衡混合DNA的分析, 其原理是当混合样本DNA主要组分的DIP基因型为缺失(S), 而次要组分的DIP基因型为插入(L)时, 用S或L引物可以对混合DNA进行有选择的扩增, 从而将次要组分与主要组分区别开。最理想的情况是主要组分与次要组分在该DIP上都是纯合的, 且等位基因的基因型不同(例如主要组分的基因型为S-S, 次要组分为L-L, 不考虑STR的多态性)。而当主要组分DNA的DIP基因型为纯合, 次要组分的DIP基因型为杂合时, 次要组分DNA只有与主要组分的DIP基因型不同的那个基因型可被检测出来[14, 16]。Oldoni等[17, 18]以模拟接触检材应用DIP-STR对混合不平衡DNA进行了鉴别。受限于实际案发现场获得的混合DNA样本的质量和数量, 现有单遗传标记扩增难以满足需要。故为了推动DIP-STR遗传标记应用于实际检案, 本研究尝试建立合适的复合扩增体系, 将单遗传标记PCR扩增转变为多遗传标记复合PCR扩增, 并减少实验次数、节约样本DNA量、降低实验成本, 以提高DIP-STR的实际可应用性。所建立的复合扩增体系经中国东北汉族人群进行了验证, 其数据又与中国西南人群、瑞士人群作了比较。
按知情同意(每位参与者都获得书面知情同意书)和匿名原则, 并经复旦大学生命科学学院伦理审查, 从中国东北吉林省长春市107名汉族无关个体采集外周血样本。
使用血液基因组DNA提取试剂盒(Leifeng, China)提取基因组DNA。根据说明书, 以NanoDrop-1000分光光度计(Thermo Fisher NanoDrop, USA)对DNA浓度定量为5 ng/μ L。
单倍型数(N)通过直接计数而得。利用Powerstats V12 (Promega)计算观察杂合度(Ho)。为评估DIP-STR辨别能力, 使用之前公布的DIP-STR有效基因型公式(I)(I = 2s2l2 + 2s3l + 2sl3)估算, 其中s和l是等位基因S和L的基因频率[14]。用t检验比较人群之间单倍型数的差异。
单遗传标记PCR实验遵循Hall等人的设计, 在含有DIP-Primer(10 μ M)0.2 μ L, STR引物(10 μ M) 0.2 μ L, FastStart Taq DNA聚合酶0.1 μ L, 快速启动Taq DNA聚合酶PCR缓冲液(10× )2 μ L, dNTP 1 μ L, ddH2O 16 μ L, 样本DNA(10 ng/μ L)0.5 μ L, 即终体积20 μ L体系中进行各单个PCR反应:95℃、5 min; 续以GeneAmp 9700热循环仪(Life Technologies)94℃、60 s, 55℃、60 s, 72℃、60 s, 共30个循环; 最后72 ℃延伸30 min。S-rs112604544-STR和L-rs112604544-STR遗传标记退火温度修改为62、52 ℃(经在50 ~65 ℃的范围内测试调整得到)。使用ABI PRISM 3730xL遗传分析仪进行毛细管电泳, 以GeneMapper V3.2软件分析电泳后的PCR扩增产物。
复合扩增体系包含多个DIP-STR遗传标记的单PCR扩增, 故要求各个遗传标记PCR反应的最适温度要彼此接近才能保证构建成功。经验证, rs112604544-STR遗传标记其S-DIP引物退火温度为62 ℃而L-DIP引物则需52 ℃, 另rs71070706-STR遗传标记三种引物也是52 ℃, 与其它遗传标记退火温度相差较大, 因而不利于构建复合基因座PCR系统, 故排除。所以本研究以其余8个遗传标记(引物设计不变)为基础, 构建复合扩增体系; 经探索PCR扩增条件及体系对取自东北地区的样本进行扩增并使用ABI PRISM 3730xL遗传分析仪毛细管电泳, 以GeneMapper V3.2软件分析复合PCR扩增产物。
复合扩增体系的分组应当满足以下条件:1)每个体系中不同遗传标记的预期长度范围不能有重叠; 2)每个体系中不同遗传标记的DIP端引物和STR端引物之间不会交叉扩增; 3)每个体系中的遗传标记数尽可能多。根据上述要求, 先依据PCR产物长度范围分析可能的组合。使用BLAST工具在NCBI序列数据库中对各遗传标记的S-DIP, L-DIP和STR引物进行比对, 以检测可能的重复序列, 确保不出现交叉扩增情况的组合。满足全部条件的组合有2种, 经过PCR扩增检验后, 确定选择实验效果较好的一种。最终复合扩增体系引物序列与设计结果见表1~2, 复合扩增PCR反应条件与体系见表3~4。
| 表1 DIP-STR遗传标记引物序列 Table 1 Primer sequences for the selected DIP-STR genetic markers |
| 表2 DIP-STR遗传标记分组情况 Table 2 Grouping of DIP-STR genetic markers |
| 表3 DIP-STR复合标记物PCR反应条件 Table 3 PCR reaction conditions for DIP-STR composite markers |
| 表4 DIP-STR复合PCR扩增体系 Table 4 DIP-STR composite PCR amplification system (respectively grouping into the following C1, C2 and C3 sets) |
将DNA样本(样本编号:DBT069)稀释至5 ng/µ L, 按照本研究已优化的实验流程进行检测, 结果如图1。
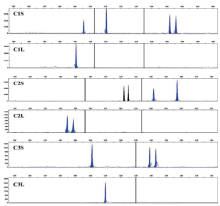 | 图1 复合扩增体系对单一DNA样本检验结果Fig.1 A single individual’ s DNA amplified with the composite PCR amplification system |
根据复合扩增体系中不同遗传标记的预期长度范围划定分隔线。C1组中3个遗传标记依据长度范围排列依次为rs34212659-STR、rs142543564-STR和rs 35032587-STR, 分隔在203 bp (rs34212659-STR L-10)和235 bp (rs142543564-STR L-11)处; C2组中依次为rs146332920-STR、rs2308142-STR和rs111478323-STR, 分隔在197 bp (rs146332920-STR L-11)和234 bp (rs2308142-STR L-14)处; C3组中依次为rs724068 28-STR和rs145423446-STR, 分隔在230 bp (rs724068 28-STR L-13)处。由图1可见, 各遗传标记峰高均匀, 峰分布清晰, 相邻的杂合峰也能很好地分开, 且非特异性扩增的峰值较低, 不影响正常扩增峰值的读数, 证明所建复合扩增体系作为取代原10个遗传标记测试组的优化是可行的。
将两份参考DNA样本(样本A:编号DBT059; 样本B:编号DBT060)稀释至5 ng/µ L, 配制测试样本1 (A∶ B=10∶ 1=5 ng∶ 0.5 ng)和2 (A∶ B=1∶ 10=0.5 ng∶ 5 ng), 按照实验流程进行检测。结果显示, 尽管微量组分在总样本量中所占的比例低于10%, 且总量不超过0.5 ng, 但PCR扩增还是能够将微量和大量组分区分开。根据结果, 样本A在C1S组中的标记物rs35032587-STR和rs142543564-STR上各有一个明显的峰, 而标记物rs34212659-STR则是空白; 样本B在标记物rs35032587-STR、rs142543564-STR和rs34212659-STR上均有一个明显的峰, 可以直观地看出A和B在标记物rs34212659-STR上是不同的。样本B在标记物rs34212659-STR的DIP单倍型上至少有一个S-DIP, 而A则一定是L-DIP。故当样本A和B以10∶ 1的比例混合时, 样本A成为了大量组分, B则是微量组分。如果A和B是一个混合斑中的两个组分, 在A已知的复合扩增体系做具体分型时, B可能的分型就能求知。这在法医物证学的实际应用中有着极高的价值, 通过确认组分B的某些标记物分型, 就能排除掉部分嫌疑人, 从而可大大缩小搜索范围。
本研究通过大量实验探索建立了合适的DIP-STR复合扩增体系, 接着又对其以中国东北地区的107位汉族不相关个体进行了验证。相关法医学参数见表5, DIP-STR遗传标记单倍型及基因频率见表6。
| 表5 从中国东北汉族人群获得8个DIP-STR遗传标记的法医遗传学参数 Table 5 Forensic genetic parameters of 8 DIP-STR genetic markers obtained from Han population in Northeast China |
| 表6 中国东北汉族人群中8个DIP-STR标记物的单倍型及基因频率 Table 6 Haplotypes and gene frequencies of 8 DIP-STR genetic markers in northeast China’ s Han population |
从表中可知:中国东北汉族人群的单倍型数目(N)在8至15之间, rs146332920-STR遗传标记的单倍型最多, 有15种, rs34212659-STR遗传标记的单倍型最少, 共9种; 观察杂合度在0.18到0.54之间, 其中rs142543564-STR遗传标记的杂合度最低, 为0.18; 而rs34212659-STR遗传标记杂合度最高, 为0.54。较低的杂合度表明, 此8个DIP-STR遗传标记适合在中国东北汉族人群中进行鉴别。中国东北汉族人群的有效基因型I值在0.24到0.37之间, 其中rs2308142-STR遗传标记的I值最小, 为0.24, rs111478323-STR和rs72406828-STR遗传标记I值最大, 为0.37。根据107个样本估计等位基因频率, 此8个DIP-STR遗传标记具有较高的有效基因型概率(I值), 这表明它们在中国东北汉族人群中具有较高的应用价值。
本研究将实验所得中国东北汉族人群的数据与2015年Hall等人发表的瑞士人群的实验数据、2017年张林等人发表的中国西南汉族人群的实验数据进行了对比[15, 19]。
中国东北汉族人群与瑞士人群单倍型数的差异比较, 采用t检验, 差异无统计学意义(P=0.193> 0.05)。中国东北汉族人群杂合度的整体水平要低于瑞士人群。中国东北汉族人群、瑞士人群的I值分别在0.24~0.37与0.25~0.36之间, 平均值为0.32和0.33。中国东北与西南汉族人群单倍型数的差异比较, 采用t检验, 差异无统计学意义(P=0.150> 0.05)。中国东北汉族人群杂合度的整体水平要低于中国西南汉族人群。中国东北与西南汉族人群的I值分别在0.28~0.37与0.27~0.37之间, 平均值为0.34和0.27。
DIP-STR复合遗传标记的设计初衷是针对混合样本的检测, 以实现经由两种遗传标记同时PCR对混合样本进行分型。但早期实验方法和操作步骤过于繁杂, 不适于平台化、规模化的法医学检测, 因此笔者进行了一系列的研究, 着力于简化操作步骤、精简试验组流程和改进实验系统, 以期在实践中能够更好地推广DIP-STR遗传标记的应用。原始方法与改进实验方法的比较如表7。
| 表7 原始方法与改进实验方法的比较 Table 7 Comparison between original methods and the improved experimental ones |
对比结果显示:与原来的十标记物测试组相比, 新的复合扩增体系将操作次数和实验成本减少了三分之二, 样品DNA消耗量减少了约70%, 这对于珍贵的法医样本具有重要意义。综上所述, 实验系统的改进是推动DIP-STR复合型遗传标记在法医实践中应用的第一步, 虽然这离DIP-STR真正投入实用尚有一段距离。目前, 这种新型遗传标记存在以下问题:1)作为一种复合遗传标记, DIP-STR测试组仍不够简化, 较之于法医学检测中大规模应用的如Y-filer等试剂盒可在复合扩增体系的基础上一次PCR实验即能检测所有基因座, 确有很大差距。如果能对DIP-STR复合扩增体系进行遗传标记东亚化的优化, 并针对实验体系继续改良至只需2次实验(L-DIP测试组和S-DIP测试组)即可获得全部分型, 那将会有更好的应用前景。2)应基于中国人群的基因库筛选更多合适的DIP-STR遗传标记, 使得DIP-STR区分能力更佳, 开发出更理想的DIP-STR法医学试剂盒, 使其适用于实际检案。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|


