作者简介:杨鑫(1982—),男,甘肃兰州人,硕士,主检法医师,研究方向为法医遗传学。E-mail:lz_yangxin@163.com
目的 对青海、甘肃境内518名藏族无关个体和平原地区134名汉族无关个体EPAS1和EGLN1基因的10个SNP位点做遗传多态性分析。方法 使用SNaPshot®多重分析试剂盒分析藏汉人群rs12907901、rs13419896、rs150877473、rs1562453、rs186996510、rs2491406、rs2491407、rs4953360、rs4953361、rs7589621共10个SNP位点的基因型、等位基因频率等遗传学参数。结果 根据基因型频率分析,汉族群体上述位点所有基因型频率符合Hardy-Weinberg平衡,藏族人群rs150877473、rs1562453、rs186996510、rs7589621共4个位点不符合Hardy-Weinberg平衡( p<0.05),同时两组人群rs13419896、rs1562453、rs186996510、rs7589621、rs4953360共5个位点存在显著差异;根据等位基因频率分析,藏汉人群在rs150877473、rs1562453、rs186996510、rs7589621共4个位点存在显著差异。结论 本研究验证了EPAS1、EGLN1基因在藏汉群体间的差异性,为SNP分子遗传标记应用于刑事科学实践提供了理论支持。
Objective To study the genetic polymorphism of 10 SNPs in genes EPAS1 and EGLN1 among 518 unrelated Tibetan individuals from Gansu and Qinghai areas and 134 unrelated Han individuals from 500-meter altitude regions.Methods 10 SNP markers (rs12907901, rs13419896, rs150877473, rs1562453, rs186996510, rs2491406, rs2491407, rs4953360, rs4953361, rs7589621) in genes EPAS1 and EGLN1 were selected to analyze their genetic information such as genotype frequency and allele frequency through SNaPshot® Multianalysis kit.Results According to genotype frequency analysis, no deviation was detected from the Hardy-Weinberg equilibrium in Han population, yet 4 SNPs (rs150877473, rs1562453, rs186996510, rs7589621) in Tibetan population to deviate from the Hardy-Weinberg equilibrium ( p<0.05). Meanwhile, significant differences were also observed in the distribution of genotype frequency in 5 SNPs (rs13419896, rs1562453, rs186996510, rs7589621, rs4953360) between the Tibetan and Han populations. The allele frequency analysis showed that there were significant differences in 4 SNPs (rs150877473, rs1562453, rs186996510, rs7589621) between the two populations.ConclusionsThis research verified the previous report that there was significant difference between Tibetan and Han populations in EPAS1 and EGLN1 SNPS genes, providing a further support for these SNP genetic markers to be used in the future forensic practice.
藏族作为世界高原人群中居住海拔最高、适应历史最长的群体, 主要分布于尼泊尔、印度、巴基斯坦、不丹及中国的西藏、青海、甘肃、四川、云南等省。据全国第五次人口普查数据显示, 中国现有藏族总人口为628 2187人, 是全国第三大少数民族。近年来, 国内外很多重要研究发现高原藏族人群EPAS1及EGLN1基因中多个SNP位点连锁单倍型及频率与平原地区汉族人群存在显著差异[1, 2, 3, 4]。与此同时, 法医遗传学者不断探讨利用特定功能基因中的特异性分子标记, 鉴别未知个体的民族属性或地域来源[5, 6, 7, 8], 从而可为刑事侦查、安全防控等工作提供参考或理论依据。
本文通过分析甘肃、青海两省藏族聚居区藏族无关个体和平原地区汉族无关个体EPAS1和EGLN1基因的10个SNP位点的多态性, 研究上述SNP位点在藏汉人群间存在的差异, 为以后利用SNP遗传位点多态性差异对相关个体的民族属性或地域来源推断提供理论依据。
收集青海果洛藏族自治州(E99° 73′ , N33° 40′ , H4017m)与海北藏族自治州(E101° 33′ , N37° 22, H3644m)、甘肃甘南藏族自治州玛曲县(E100° 77′ , N34° 87′ , H3835m)和天祝藏族自治县(E103° 01′ , N36° 48′ , H2178m)共2个省份4个藏族聚居区的藏族人群共518个无关个体的血样, 选取海拔低于500米的平原(山东、河南、安徽3省)汉族人群134位无关个体作对照。
美国Thermo Fisher的高速离心机、平板高速离心机、振荡器、PCR扩增仪、测序仪、; 美国Bio-Rad的凝胶成像仪; 美国Roche 的DNA聚合酶; 北京TIANGEN的血液基因组DNA提取试剂盒; 美国Qiagen的血卡样本基因组DNA提取试剂盒; 美国Thermo Fisher的 SNaPshot® 多重分析试剂盒。
全血样本DNA采用血液基因组DNA提取系统提取, 血卡样本DNA采用Investigator Kit提取。提取产物终浓度在20 ng/µ L以上。
选取文献报道[9, 10, 11, 12, 13]且存在显著性差异的SNP位点rs12907901、rs13419896、rs150877473、rs1562453、rs186996510、rs2491406、rs2491407、rs4953360、rs4953361、rs7589621共10个(见表1)行研究。
| 表1 SNP位点信息汇总表 Table 1 Information of selected SNPs |
依据NCBI数据库内上述位点相关序列, 使用Primer6.0软件设计引物, 引物要求具有特异性, 片段长度存在差异, PCR反应的退火温度范围集中但略有差异。
根据扩增片段的长度, 将10个SNP遗传标记按照SNaPshot® 多重分析试剂盒设计要求, 进行分配并构建多重PCR体系。为了改变引物的长度, 区分不同的位点, 在引物的5’ 端添加GACT为重复单元的单碱基重复序列尾巴; 根据dT数量的差异, 每个相邻检出位点的SNP引物碱基数差异保持在3~8个碱基之间。SNP位点及SNaPshot引物信息参见表2。
| 表2 SNaPshot引物信息汇总表 Table 2 Information of SNaPshot primers |
检测EPAS1、EGLN1基因上述SNP位点在134名平原汉族无关个体以及518名青海、甘肃藏族无关个体中的基因型, 计算各SNP位点等位基因频率、基因型等信息, Hardy-Weinberg遗传平衡检验、连锁不平衡原汉族人群。
通过对134名平原汉族无关个体以及518名青海、甘肃藏族无关个体EPAS1、EGLN1基因10个特异SNP位点的分型检验和分析, 其基因型及等位基因频率分布等群体遗传学参数见表3。可以看出, 汉族人群10个SNP位点基因型分布频率介于1~112之间(n=134), 所有10个位点均符合Hardy-Weinberg平衡; 而藏族人群相应10个SNP位点的基因型分布频率介于58~256之间(n=518), 其中有rs150877473、rs1562453、rs186996510、rs7589621共4个位点不符合Hardy-Weinberg平衡(p< 0.05)。
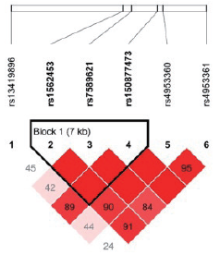
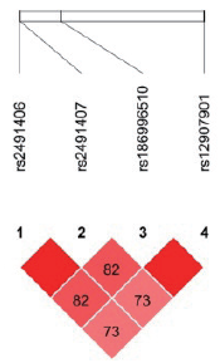
在134名汉族个体的10个位点中, 有5个位点的特定基因型检出率不足10 %, 分别为rs150877473(GG, n=1)、rs186996510(CC, n=1)、rs1562453(TT, n=2)、rs7589621(GG, n=9)、rs4953360(AA, n=6); 而藏族人群样本中尚未发现基因型检出率不足10 %的位点。此外, rs2491406和rs2491407两个位点均位于EGLN1基因中, 两个位点相隔263个碱基, 呈高度连锁遗传, 故基因型频率完全一致。藏汉人群上述两个基因SNP位点连锁分析结果见图1~4。
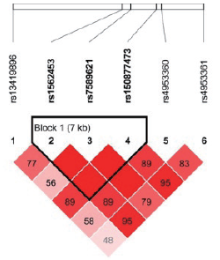 | 图1 藏族EPAS1 6个SNP位点连锁不平衡分析Fig.1 Linkage disequilibrium among the 6 SNPs of EPAS1 in Tibetan population |
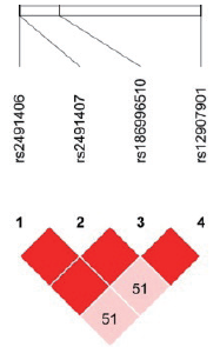 | 图2 藏族EGLN1 4个SNP位点连锁不平衡分析Fig.2 Linkage disequilibrium among the 4 SNPs of EGLN1 in Tibetan population |
由表4可以看出, 藏汉人群EPAS1、EGLN1基因10个SNP位点等位基因频率分布也存在差异, 其中汉族人群10个SNP位点等位基因分布频率介于0.0858~0.9142之间(rs150877473、rs186996510), 而藏族人群10个SNP位点等位基因分布频率介于0.3089~0.6911之间, 多态性及正态分布均衡性高于汉族人群。
分析表3中藏汉人群EPAS1、EGLN1基因10个SNP位点基因型频率的差异发现, rs13419896、rs1562453、rs186996510、rs7589621、rs4953360共5个位点的基因型频率存在统计学差异, 这一结论与表4中等位基因频率差异的结论相似, 后者有rs150877473、rs1562453、rs186996510、rs7589621共4个位点的等位基因频率在藏汉人群间存在差异。值得注意的是, 等位基因频率存在差异的4个位点全部为藏族人群Hardy-Weinberg检验不平衡的位点。
| 表3 藏汉人群相关SNP位点基因型统计表 Table 3 Genotype frequencies of SNPs in Tibetan and Han populations |
| 表4 藏汉人群相关SNP位点等位基因频率统计表 Table 4 Allele frequencies of SNPs in Tibetan and Han populations |
EPAS1(Endothelial PAS Domain Protein 1)基因, 又称为HIF-2α , 定位于人染色体2p16-21, 含有16个外显子, 序列长度约90kb。该基因在低氧条件能够明显提高细胞内HIF-2α 蛋白含量, 其机制是HIF-2α 蛋白内存在一种依赖氧压的降解域, 在常氧条件下HIF-2α 蛋白质能够迅速地降解, 而低氧能够提高HIF-2α 蛋白质的稳定性, 使其含量升高。EGLN1(egl nine homolog 1)是调节HIF-1α 的一个关键分子, 它能够直接感受氧分压, 是一种双加氧酶的氧感受器, 通过催化HIF脯氨酸残基发生羟化反应介导其降解。上述两个基因是HIF通路中的重要基因, 在人对低氧环境的调节通路中起到核心作用, 与缺氧应激、急慢性高山病等均存在显著关联[9, 10]。
近年来, 国内外很多重要研究发现高原藏族人群EPAS1及EGLN1基因中多个SNP位点连锁单倍型及频率与平原地区汉族人群存在显著差异。Peng[2]研究了7个藏族人群共1334名个体的EPAS1/EGLN1基因SNP位点后, 发现藏族人群EPAS1基因rs13419896(A; 藏族0.54~0.86, 汉族0.28, 日本人0.39)、rs4953354(G; 藏族0.42~0.83, 汉族0.13, 日本人0.12)、rs1868092(A; 藏族0.33~0.81, 汉族0.08, 日本人0.09)和rs2275279(T; 藏族0.53~0.74, 汉族0.32, 日本人0.24)共4个SNP位点在频率上与平原汉族及日本人有显著性差异。Xiang[11]对46名藏族无关个体EGLN1基因中相关SNP分析发现, 藏族在位点rs186996510的非同义突变(C-G)频率与平原人群存在显著差异, 不同地区藏族人群中C-C基因型的频率介于0.6429~0.7583, 而中国汉族、日本人、欧洲人及非洲人该基因型频率分别只有0.0103、0.0056、0.0059、0.0227; 类似的结论也存在于位点rs12097901。柯金坤[12]分别选取海拔4454 m的西藏浪卡子县藏族、海拔3100 m的青海贵南县藏族、海拔2500 m的云南贡山县藏族及山东平原汉族EPAS1基因14个SNP位点进行分析, 发现rs1562453位点的等位基因频率在藏汉人群之间存在显著差异, 而rs4953361位点的等位基因频率虽然在云南藏族与汉族人群之间差异不显著, 但在西藏藏族、青海藏族与汉族人群间存在显著差异, 显示长期高海拔环境对世居人群EPAS1基因有选择作用, 并可造成藏汉人群在相关基因SNP位点频率及多态性上存在稳定遗传的差异。
根据基因型频率分析, 汉族群体上述位点所有基因型频率符合Hardy-Weinberg平衡, 藏族人群rs150877473、rs1562453、rs186996510、rs7589621共4个位点不符合Hardy-Weinberg平衡(P< 0.05), 同时两组人群rs13419896、rs1562453、rs186996510、rs7589621、rs4953360共5个位点存在显著差异; 根据等位基因频率分析, 藏汉人群在rs150877473、rs1562453、rs186996510、rs7589621共4个位点存在显著差异。这些基因型及等位基因频率方面的差异验证了前期Xiang[11]、柯金坤[12]、Yi[13]等人的相关研究中关于藏汉人群在EPAS1、EGLN1基因中存在差异的事实, 其相应位点在藏族人和平原汉族之间的差异相似或接近, 由此说明这些基因及其SNP位点在藏汉人群间的差异性是客观、稳定的[14, 15, 16, 17, 18]。这一结论为从生物学角度研究人类在极端环境(高原和高山)下进化及遗传适应性提供了条件。而且, 这些差异的深入研究和探索, 极有可能为推断相关人员的藏民族属性或具体地域来源提供指向性线索, 具有重要的研究价值和应用前景。
本研究在样本数量、SNP位点数量及特异性选择方面的工作还需加强, 特别是通过这些SNP位点区分汉藏人群民族属性方面仍有很多工作要完善。法医物证鉴定中关于个体身份辨识能力的诸多研究指出, 要达到现有AmpFlSTR Identifiler 试剂盒鉴别个体身份的水平, 其相应的SNP位点使用数量至少需达到或超过38~43个, 且这些SNP位点必须满足杂合度高(≥ 0. 4)、不同人群间等位基因频率差别小( Fst< 0. 06) 及遗传标记间没有显著的连锁状态这三个要求[19, 20, 21]。由此可见, 本文的研究还需在以后重点加强两方面的研究, 一是筛选和验证更多特异性的SNP位点, 如rs1868092[9]、rs480902[9]、rs4953354[16]、rs13428739[22]等; 二是建立基于系统进化理论的数学模型或统计学公式。这样利用EPAS1、EGLN1基因特异SNP位点鉴别藏汉人群民族属性的能力和证据强度将会大幅提升。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|