作者简介:宋炳轲(1987—),女,河南许昌人,硕士研究生,主要从事法医遗传学的研究。Tel:010-66269472; E-mail:sbk1987@126.com
目的 利用叶绿体上的间隔区psbA-trnH条形码序列鉴别毒品原植物大麻及其混伪品,为毒品原植物大麻的鉴定提供新方法。方法 采用PCR法扩增psbA-trnH间隔序列,双向测序后运用 CodonCode Aligner、MEGA5.1软件进行数据处理,构建系统聚类树(NJ树)。结果 psbA-trnH条形码序列分析表明大麻种内与种间遗传距离具有较大差异,基于psbA-trnH条形码构建的NJ树可鉴别大麻及其混伪品。结论 psbA-trnH序列可以作为鉴定毒品原植物大麻及其混伪品的候选条形码序列,为毒品原植物大麻的快速、准确鉴定提供了新方法;DNA条形码技术在犯罪案件中涉及的植物鉴定中具有极大的应用前景。
Objective The psbA-trnH sequence of cpDNA was utilized as a novel technique to discriminate drug original plants of cannabis and adulterants. Methods The psbA-trnH regions were amplified and sequenced bi-directionally. Then the obtained sequences were assembled using the CodonCode Aligner. The psbA-trnH genetic distances of the regions were computed in accordance with the kimura 2-parameter (K2P) model and Neighbor-joining (NJ) phylogenetic trees were constructed using MEGA5.1.Results The psbA-trnH sequences analysis showed that K2P distances of inter-and intra-specific were different significantly. The NJ trees showed that the cannabis could be distinguished easily from adulterants. Conclusions The psbA-trnH fragment could be used as a candidate barcode for identifying cannabis from adulterants and it provide a new method to accurately identify cannabis. DNA barcoding has broad prospects for plant identification in criminal investigation.
大麻为大麻科大麻属1~2年生雌雄异株的草本植物。由于大麻植物所含的四氢大麻酚(THC)具有致幻作用, 所以大麻被列为与海洛因、可卡因并列的3大毒品之一[1]。在高额利润的驱使下, 全球范围的大麻种植和非法交易活动屡禁不止。与此同时, 在大麻犯罪案件中缴获成批未加工的大麻植物(如整株植物、种子等)的情况也屡见不鲜; 而且在贩毒案件中犯罪分子经常用烟草、桑叶及经济植物小麦等掺在毒品原植物大麻中来掩盖毒品进行走私和贩卖; 另外在毒品走私案件中经常还会涉及到罂粟, 它是制取鸦片的主要原料并且比大麻的毒性更强; 而且由于罂粟的花型、颜色及果实与同科属观赏植物虞美人[2]极为相似, 因此仅从外观形态上很难鉴别。故如何科学地将涉毒案件中犯罪分子精心伪装的毒品及毒品种类与其他易混植物进行区分和鉴别, 是公安机关缉毒取证的难题和关键。在法庭科学中, 虽然可用传统的形态学方法对大麻进行鉴定, 用理化方法如常规化学法、薄层色谱法等对大麻毒性成分进行测定, 但这些方法都有很大的局限性, 对检材的要求较高且很难或根本无法区分, 所以为了有效的打击大麻毒品犯罪, 法庭科学急需寻找可以快速准确对大麻进行种属鉴定的新方法。
2003年加拿大学者Hebert等利用线粒体细胞色素C氧化酶亚基I(Cytochrome C oxidase sub-unit I, COI)中的一段650bp序列, 发现该基因序列在动物界不同分类水平上都具有良好的识别能力。从而提出了DNA条形码(DNA barcoding)的概念。建立了一种新的物种鉴定技术[3], 为解决传统分类学面临的难题提供了新方法。由于线粒体C01基因在植物中的进化速率远慢于动物, 因此很多学者认为植物的条形码应从进化速率较快的叶绿体基因组中选择[4, 5]。psbA-trnH序列是进化速率最快的叶绿体间隔区之一, 两端存在75 bp的保守序列, 便于引物的设计。作为植物DNA条形码的候选片段。trnH-psbA序列具有引物通用性较好、扩增成功率较高[6, 7, 8, 9]等特点, 并且平均长度较短, 有利于对降解材料的扩增。故在综合全面考虑后本文最终选择trnH-PsbA作为候选条形码序列来对毒品原植物大麻及其易混品进行鉴定。
采自新疆玛纳斯地区的大麻(cannabis sativa L.)叶片样本6份、大麻仔6份, 一个新疆涉毒案件中缴获的大麻烟土样本若干; 罂粟(Papaver_somniferum_L)、虞美人(Papaver rhoeas)、烟草(Nicotianatabacum)、桑叶(Morus alba L.)和小麦(TriticumaestivumLinn)标本各6份(均由公安部物证鉴定中心提供)。
1.2.1 DNA 提取及定量 取干叶10mg, 利用植物DNA提取试剂盒(Tiangen Biotech Co.中国)提取大麻全基因组DNA, 用Nanodrop1000核酸定量仪进行定量, 定量结果见表1。
| 表1 大麻及其混伪品样本全基因组DNA定量结果 |
1.2.2 PCR扩增与琼脂糖凝胶电泳 采用叶绿体DNA的通用引物psbA-trnH[10]进行扩增(引物序列见表2, 由上海英潍捷基公司合成), 扩增反应在eppendorf扩增仪上进行, PCR反应总体系为50μ L, 内含2× PCR Master mix 25μ L, 正反向引物(10mmol)各1μ L, 去离子水23μ L, 模版DNA1μ L。PCR反应程序为:94℃预变性5min; 94℃变性1min, 55℃退火1min, 72℃延伸1.5min, 共30个循环; 72℃终延伸20min。PCR产物用1%琼脂糖进行凝胶电泳检测。
| 表2 psbA-trnH通用引物序列 |
1.2.3 扩增产物测序 对扩增产物进行双向测序, 测序反应由上海生物工程公司完成。
1.2.4 序列分析 对测序得到的峰图利用CodonCode Aligner V4.0.3(CodonCode Co.美国)进行比对和拼接, 去除引物区, 获得psbA-trnH序列。将得到的序列利用MEGA5.1进行多重比对, 基于Kimura-2-parameter(K2P)双参数模型计算遗传距离, 并用邻接法(neighbour-joining)构建系统聚类树(NJ 树), 同时以 bootstrap(自展支持率1000次)重复检验各分支的支持率。
实验结果表明psbA-trnH对6份大麻叶片及大麻仔和大麻烟样本、6份罂粟、虞美人、烟草、桑叶和小麦的的扩增及测序成功率均为100%。
经过序列比对发现采自新疆地区的大麻叶片样本、大麻仔样本及在新疆地区案件中缴获大麻烟土样本的psbA-trnH引物扩增区域测序所得的序列长度均为404bp, 且这些样本的序列完全一致, 未发现变异位点。
将罂粟与大麻进行序列比对, 比对后长度为序列长度为335bp, 有80个碱基变异, 92个碱基插入或缺失; 烟草与大麻比对后序列长度为515bp, 有104个碱基变异, 115个碱基插入或缺失; 桑叶与大麻比对后序列长度为605bp, 有220个碱基变异, 89个碱基插入或缺失; 小麦与大麻比对后序列长度为636bp, 有135个碱基变异, 128个碱基插入或缺失; 虞美人与大麻比对后序列长度为324bp, 有74个碱基变异, 127个碱基插入或缺失。将罂粟与虞美人进行序列比对, 比对后序列长度为335bp, 有32个碱基变异, 6个碱基插入或缺失。
大麻、罂粟、烟草、虞美人、桑叶和小麦的种间K2P距离如表3所示, 可以看出大麻与其他伪混品的种间遗传距离平均为0.3616。大麻与虞美人的遗传距离最小为0.299, 与小麦的K-2p遗传距离最大为0.5161。新疆大麻种内遗传距离原小于大麻的种间遗传距离。
| 表3 大麻及其混伪品的种内及种间K2P距离 |
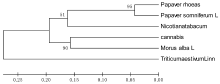
基于psbA-trnH条形码, 通过邻接法构建NJ树。从图1中可以看出, 基于psbA-trnH的系统聚类树中大麻与桑叶聚在一起, 自展支持率为90%, 这是因为大麻和桑叶同属于桑科植物; 罂粟和虞美人能够聚在一起, 其自展支出率为95%, 这是因为罂粟与虞美人同属于罂粟科植物; 小麦自成一支表现了很好的单系性。基于此聚类分析表明, psbA-trnH条形码序列不仅能够将大麻与混伪品植物区分开来; 而且能够把毒品犯罪活动中最常见的两种毒品原植物大麻和罂粟鉴别开来; 另外罂粟和虞美人这两种从形态学上很难区分的植物也可以利用DNA条形码技术进行区分, 这些对于打击毒品走私犯罪活动有极大的应用价值。
毒品问题诱发大量的违法犯罪活动, 严重影响社会稳定, 危害国家的安全和和谐社会的构建, 已成为世界范围内的一大社会公害。近些年来我国毒品消费市场持续扩大, 毒品滥用种类越来越多, 毒品问题呈蔓延之势, 危害日趋严重。打击毒品犯罪一直是公安机关乃至全社会的重要责任。在我国, 大麻和罂粟是常见的制毒贩毒的原材料, 也是毒品犯罪活动中最常见的两种毒品原植物, 如果能对大麻和罂粟进行种属鉴定, 这不仅可以帮助警方追查贩毒集团或经销商的贸易路线, 还可以帮助确定有大麻或罂粟植物的犯罪现场及毒品的类型达到准确判罪的目的。DNA条形码技术操作简便、易于实现, 并且鉴定准确性高、重复性好。Kress 等[3]分析的53科80属99种植物中psbA-trnH的扩增长度为247~1221bp, 扩增成功率达到100.0%, 在所选取的10个片段中序列属水平差异度最高, 99种植物均具有独特的间隔序列, 符合理想条形码标准。Kress 等[7]以及 Fazekas 等[8]的研究结果也表明psbA-trnH在备选片段中的扩增成功率和物种正确识别率都是最高的。故本研究分析了毒品原植物大麻及其混伪品烟草、虞美人、桑叶和小麦及罂粟的叶绿体上的psbA-trnH序列的变异情况和K2P距离, 并基于K2P距离建立了系统进化树, 表明毒品原植物大麻及其混伪品的psbA-trnH条形码序列具有较大差异, 基于psbA-trnH构建的 NJ 树聚类分支明显均证明psbA-trnH序列可以准确鉴定毒品原植物大麻及其混伪品, 为毒品原植物大麻的鉴别提供了新的分子水平的研究方法。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|