作者简介:宫荣彬(1974—),男,安徽淮北人,副主任法医师,理学学士,主要从事法医物证的检验和研究。Tel:0551-2801663; E-mail:grbhb@163.com
目的建立连接酶链反应(Ligase Chain Reaction,LCR)应用于法医学领域的初步方法探索。方法选择人Amelogenin基因座进行连接酶链反应。针对人染色体Amelogenin基因座Xp22.1-.3、Yp11.2上M55418、M55419的序列,自行设计特异引物,建立LCR最佳体系,对男性和女性Amelogenin基因座进行分型。结果巢式LCR能够对男性和女性Amelogenin基因座正确分型。结论LCR技术能够应用于人Amelogenin基因座分型,但尚需进一步简化和改进。
Objective To explore the forensic application of Ligase Chain Reaction (LCR).Methods The human Amelogenin locus was selected for LCR. According to the sequences of M55418 on the human Amelogenin chromosome loci Xp22.1-3, and M55419 on Yp11.2, the primers was designed and the LCR system was set up for genotyping male and female.Results The genotypes of male and female Amelogenin loci can be get by nested LCR correctly.Conclusion LCR can be applied to human Amelogenin locus genotyping.
连接酶链反应(Ligase Chain Reaction, LCR)是继聚合酶链反应(polymerase chain reaction, PCR)之后发展起来的一项具有良好应用前景的DNA扩增技术[1]。它在热稳定的DNA连接酶的作用下, 通过连接与模板DNA互补的两个相邻寡核苷酸链, 能够快速进行DNA片段扩增。若相邻两个寡核苷酸接头处与靶核苷酸之间即使有一个碱基错配, 也不能被连接和扩增。
目前, LCR因其应用于DNA扩增, 同时能够区别单个位点的碱基突变, 在国内外医学领域得以发展, 在诊断遗传性疾病和某些感染性疾病(如病毒或支原体感染)上取得了很好的效果[2, 3, 4, 5], 同时为分子生物学研究提供了新的技术发展方向。LCR能否应用于法医学领域, 本文针对较容易鉴别的人血样Amelogenin基因进行检验, 做了初步的研究, 报道如下。
(1)未知生物检材:来源于实际案件中的现场检材48份, 分别采用Chelex法或有机溶剂法提取DNA。(2)已知样本:取6名男性和两名女性的血液各200μ L, 用Chelex法提取DNA。(3)对照样本:男性样本9948, 1ng/μ L; 女性样本9947A, 1ng/μ L; 阴性对照:水。
TaqDNA连接酶(纽英伦生物技术北京有限公司), TaqDNA聚合酶、dNTP(上海生工), GENMED超声化鲑鱼精单链DNA(sssssDNA, 上海杰美基因医药科技有限公司)。
引物设计:主要针对Amel X、Amel Y即M55418、M55419的序列。分别为(5’ -3’ ):引物1:GTTGATTCTTTATCCCAGAT; 引物2:ATCTGGGATAAAGAATCAAC(5’ 磷酸化); 引物3:GTTTCTCAAGTGGTCCTGAT(5’ 磷酸化); 引物4:ATCAGGACCACTTGA GAAAC(5’ 标记FAM); 引物5:ACATTCTTCATCCCAAATAAAGTG; 引物6:CACTTT ATTTGGGATGAAGAATGT(5’ 磷酸化); 引物7:GTTTCTCAAGCGGTCCCAAT(5’ 磷酸化); 引物8:ATTGGGACCGCTTGAGAAAC(5’ 标记FAM); 引物9:CCCTG GGCTCTGTAAAGAA; 引物10:ATCAGAGCTTAAACTGGGAAGCTG。
2.1.1 扩增体系 在10μ L体系中, 含有20mmol/L Tris-HCl (pH7.6)、25mmol/L KAc、10mmol/L Mg(Ac)2、10mmol/L DTT、1mmol/L NAD、0.1%Triton X-100、400ng/μ L sssssDNA、10mmol/L引物1-8的混合液、8U TaqDNA连接酶、已知样本/对照样本各1μ L。
已知样本经AB7500型实时荧光PCR定量仪定量, 配制成1ng/μ L 和50 ng/μ L, 分别进行扩增。
2.1.2 扩增条件 95℃预变性5min, 94℃1min, 59℃4min, 共36个循环。
2.1.3 检测 使用AB 3100型测序仪进行荧光毛细管电泳检测。
2.2.1 技术路线 先利用PCR获得大量的DNA短链, 其包含需要连接的目的片段。然后再进行LCR, 判断分型。
2.2.2 PCR 用引物9、引物10对已知样本和对照样本进行扩增, 条件为:95℃11min, 94℃30 s, 59℃30 s, 70℃30 s, 共24个循环, 60℃30min, 4℃保存。
2.2.3 LCR 对上述PCR产物进行LCR扩增。扩增体系、扩增条件和检测方法均同直接LCR。
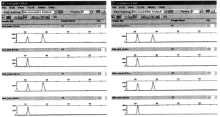
在直接LCR检测中, 各已知样本和对照样本均无产物; 在巢式LCR检测中, 各已知样本和对照样本均能正确分型, 男性样本呈两条、女性样本呈一条产物峰, 阴性对照无产物峰(见图1)。
48份案件未知样本用巢式LCR进行检测, 结果准确率100%。
巢式LCR检测的灵敏度:取男性样本9948(1ng/μ L)、女性样本9947A(1ng/μ L)稀释至0.01ng/μ L, 结果均能正确分型。
LCR是近年来发展的一项新技术, 由于其在单位点突变检测方面较PCR更加准确, 在医学诊断上有很高的灵敏度和特异性[2, 3, 4, 5], 如沙眼衣原体、淋病奈瑟氏菌等等。其反应原理见图2。
牙釉质蛋白基因(amelogenin)位于X染色体Xp22、Y染色体Yp11.2, 编码原牙釉质蛋白, 在GenBank中分别为M55418、M55419的序列。LCR分型的依据在于M55418较M55419缺失6个碱基。
本研究表明, 直接LCR对1ng至50ng的DNA检测结果均为阴性。巢式LCR获得清晰准确的检测结果。比较显示出, LCR扩增中每个循环连接反应的平均效率较低。连接效率可以通过改变缓冲液的条件加以提高, 如用NH4Cl, MnCl2, 多聚胺类, 聚乙二醇, 或改变酶的含量以及热循环的次数和温度, 但是根本因素在于连接酶的连接活性[6, 7, 8, 9, 10, 11, 12]。因此, 直接LCR用于法医检案, 从连接反应上还必须进一步寻找更佳的连接酶, 提高连接的平均效率, 提高检测的灵敏度。
在本研究中, 巢式LCR检测的灵敏度和特异性很高, 其灵敏度可以达到0.01ng/μ L。在极微量、低拷贝(LCN)模板的检测中, 巢式LCR系统可以发挥出较大的作用。本研究旨在摸索LCR技术应用于我国法医学领域的初步方法, 但是与PCR技术相比, 仍存在着操作相对复杂的缺点, 还需进一步完善和改进。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|